将目标基因克隆到质粒载体中
克隆系统和质粒载体选择
基因工程在世界各地成千上万的实验室中使用。鉴于其重要性,许多流行的DNA组分的克隆策略均未标准化是值得注意的。造成这种标准化不足的原因是,克隆试剂盒和创新技术有许多选择。 使用IP-heavy试剂盒并不总是必要的,因为使用Oxford Genetics的标准化质粒可以更容易地通过限制酶消化进行传统克隆。
Oxford Genetics的目标是设计一种DNA质粒系统,该系统可以容纳研究人员在单个质粒中可能需要的大多数功能性DNA插入物。通过优化我们的起始载体,我们系统中的每个DNA组分都可以被移除,并交换为我们预先设计和测试的数百个其他DNA区段。这就是SnapFast™的原理。我们的所有构建体都经过预先筛选,找出密码子使用不当和限制性位点相互矛盾的情况。在可能的情况下,已经去除了稀有密码子和限制性位点以实现有效表达,并确保限制性位点不限制其他SnapFast DNA区段的克隆。
我们拥有全球最大的质粒库之一,为我们提供的几乎所有插入物提供多种表达和克隆选择。我们的产品范围包括:
- 在任意位点有最大的肽标签范围可供选择(26)!
- 5种配置中共9种报告基因
- 超过20种信号肽
- 超过40种用于哺乳动物、细菌和酵母表达的启动子
- 共计10种抗生素和代谢选择方案
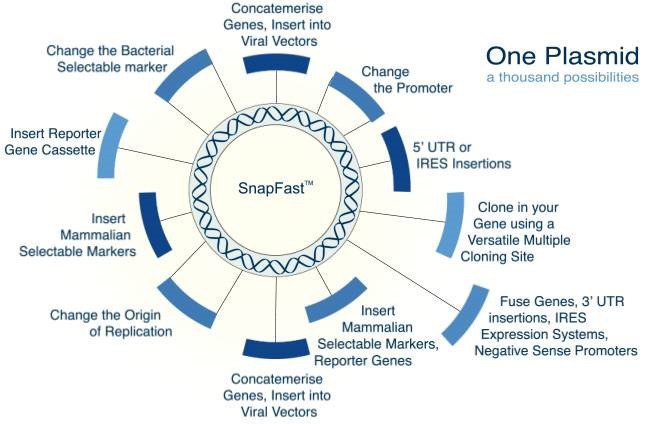
SnapFast系统的优点:
- 所有质粒均与标准克隆(LIC、InFusionHD、Gibson Assembly、Seamless Geneart)兼容。
- 1600多种独特的DNA区段可供选择并兼容。
- 轻松、高效,简单的工程策略。
- 预先设计的兼容性大大提高了成功率。
- 无插入片段大小限制。低拷贝起点提高了稳定性。
- 与许多预先存在的克隆载体和一系列穿梭载体兼容,便于基因转移。
- 从SnapFast载体能轻松克隆到一系列替代系统,包括病毒载体。
- 内置调控序列和功能(例如基因叠接、绝缘子和终止子序列)。
* 需要使用同源或TypeIIS位点进行引物设计
** 专利限制克隆技术或产品的使用
^ 在线提供的工具有助于设计
LIC = 不依赖连接酶的克隆
质粒限制性消化
制备性消化是切割DNA,以准备与另一段DNA连接,而不是简单地确认DNA的身份。您的目标应该是同时设置片段和载体的消化。这样可以节省时间。本实验方案假定在Tris-EDTA(TE)或洗脱缓冲液(EB)或无核酸酶H2O中的质粒溶液为200-300 ng/μl和约3-5 kb。较大的质粒可能需要使用更多的体积,因为在相同浓度下,质粒的拷贝数将更少。
在本特定示例中,可以执行以下操作:
10-20 μl质粒(200-300 ng/μl,总量3-5 μg)
10 μl 10X限制性缓冲液
10 µl 10X牛血清白蛋白(BSA,终浓度通常为100 μg/ml)(货号:A7906)
1.5-2 µl 限制酶1(10-20 单位/µl)
1.5-2 µl 限制酶2(选用)
用TE(货号:T9285)或无核酸酶的水(货号:W4502)定容为70或100 μl总体积
将上述试剂加入无菌1.5 ml Eppendorf中,先加入TE或水,再加入质粒/DNA,然后加入限制性缓冲液和BSA,充分混匀。最后,加入限制酶。限制酶的选用取决于您的目标和质粒图谱,但可包括EcoR I、BamH I或Hind III。在适当温度(通常为37°C,但务必确保不属例外情况,例如SwaI则用25°C)下孵育反应物,并启动闹钟(正计时)。运行反应40-60分钟。现在您可以使用碱性磷酸酶(CIP)对载体进行去磷酸化。此时应灭活限制酶,这是标准做法。
在琼脂糖凝胶上进行消化(用非常大的培养孔装载大样品),并检查线性化质粒大小的单一条带的结果。这个消化有三种可能的结果:
- 完全没起作用。无原因的消化时间长,因为反应失败了。如果酶已经在之前另一个质粒上起作用,您可以重新沉淀您正试着切割的质粒,或者在沉淀之前进行新的苯酚提取并再次尝试。否则,使用另一个质粒检查酶是否仍然可以。
- 已经起作用,但是在40分钟后载体没有被完全切割(在您所预期的近似分子量处可见多个条带)。您可以根据进度估计还需要多长时间,并尝试另一种消化。
- 完全消化。现在您就有了一个可以用于连接程序的线性质粒。
提示:限制酶对温度变化非常敏感,因此避免将其置于冰上。理想的做法是使用-20°C冷藏箱。另外,避免用手指拿着限制酶所在的试管底部,在处理酶原料时尽量快速工作。如果妥善对待它们,它们可以用得久些。
片段限制性消化
这是制备性消化的实验方案,即切割DNA,以准备与另一片DNA连接,而不是简单地确认DNA的身份。通常这些DNA片段是环状质粒,但它们也可以是PCR片段。您的目标应该是同时设置片段和载体的消化。这样可以节省以后的时间。
本实验方案假定在Tris-EDTA(TE)或洗脱缓冲液(EB)或无核酸酶H2O中的质粒溶液为200-300 ng/μl和约3-5 kb。较大的质粒可能需要使用更多的体积,因为在相同浓度下,质粒的拷贝数将更少。
在本特定示例中,可以执行以下操作:
10-20 μl质粒(200-300 ng/μl,总量3-5 μg)
7.5 μl 10X限制性缓冲液
7.5 µl 10X牛血清白蛋白(BSA,终浓度通常为100 μg/ml)(货号:A7906)
1.5-2 µl 限制酶1(10-20 单位/µl)
1.5-2 µl 限制酶2(选用)
用TE(货号:T9285)或无核酸酶的水(W4502)定容为70 μl总体积
DNA片段限制性消化实验方案
将上述试剂加入无菌1.5 ml Eppendorf中,先加入TE或水,再加入质粒/DNA,然后加入限制性缓冲液和BSA,充分混匀。最后,加入限制酶。在适当温度(通常为37°C,但务必确保不属例外情况,例如SwaI则用25°C)下孵育反应物,并启动闹钟(正计时)。运行反应40-60分钟。在琼脂糖凝胶上进行消化(用非常大的培养孔装载大样品),并检查插入片段大小的单一条带的结果。
DNA片段凝胶切除
要切除条带,您需要一把干净的手术刀或剃刀刀片。需要考虑的一个重要因素是要给DNA带多少琼脂糖。DNA与琼脂糖量的比例越高,带给连接的污染物就越少。
用于从琼脂糖凝胶中提取DNA的标准方法是用紫外光可视化溴化乙锭(货号:H5041)染色。这并不是理想的方法,因为紫外线会破坏DNA,并导致突变和降低连接效率。另一种方法是使用Clare Chemical暗场透射照明阅读器,但有时很难看到数量低的条带。 如果您有大量的DNA,这些系统则是理想选择。另外,您也可以通过在大孔附近的泳道中运行少量切割的DNA进行盲切除,并使用其位置在下一个孔中切除大量DNA。对这种技术要非常小心,因为一些条带在凝胶中可能是非常小的纵向 / 从上到下条带,如果切割不准确,就可能会错过。切除后,您可以查看剩余的凝胶是否有条带,要快一点做,因为如果您没有得到DNA,应避免紫外线破坏它。
小心对待靠得很近的DNA片段。该技术的关键是将载体骨架与片段分开以防止污染。只是因为您在切除时看不到东西,并不意味着它不在那里,您只能看到高斯分布的DNA的峰值,而且,尽管条带可能看起来是分开的,但它们可能并不是分开的,您可能从意外挨得很近的条带得到一些DNA。
提示:就分子生物学而言,这是一个相对危险的操作程序。紫外线、致癌的溴化乙锭和手术刀都极不安全。如果您不确定应采取什么必要的预防措施,则请遵守所有当地安全规则和规定,并咨询当地安全官员。
提示:考虑选择较安全的化学品来显示DNA。Nancy 520(货号:01494)和Syber Safe(货号:S9305和S9430)均显示出比溴化乙锭更低的致突变性。
凝胶碎片清除
为了从琼脂糖凝胶中用凝胶提取DNA片段,通常最值得使用是GenElute™ 凝胶提取试剂盒(货号:NA1111)之类的制备试剂盒。它们相对便宜且高效。在不使用试剂盒的情况下,还有其他一些方法,比如用透析管,在封口膜之间挤压凝胶片段以挤出溶液中的DNA,但是经常使用这些方法会导致高水平的污染物和低DNA产率。
DNA凝胶提取试剂盒通常由珠子或旋转柱组成。每个试剂盒可容纳的DNA片段大小会有所不同,因此请检查试剂盒是否可以提取您正在处理的DNA的大小范围。大多数试剂盒的范围为100bp - 5 Kb,但有些试剂盒具有更低或更高的处理能力。
离心柱和珠子试剂盒的原理是将DNA与某种物质(通常是硅土树脂)结合,然后让珠子沉淀洗去污染物,或者洗去柱子上的污染物。最后的洗脱步骤从树脂/珠子/柱子释放DNA,为您提供洁净的DNA片段。然而,即使是最好的提取试剂盒,仍然会携带一些污染物,因此,应该避免使用大量在连接中通过凝胶提取产生的溶液。当向连接添加片段时,有时“少即是多”。尽量避免使连接含有体积超过30%的凝胶提取分离的液体。
凝胶提取试剂盒的差异
我们在自己的实验室中测试了六种不同的DNA凝胶提取试剂盒,因为这个过程对我们的工作至关重要。我们发现有些试剂盒总是能以高产量产出纯DNA,有些试剂盒有时产量不错,有时就不行,而其余的则一直表现不佳。
如果从一个质粒中凝胶提取一个片段,可能应该预计到DNA的浓度(如果使用分光光度计)会很低,因为您开始会用一个大质粒(最可能约为5 kb),然后切出一个小得多的质粒亚组分(可能是1 kb),试剂盒永远不会从凝胶中纯化回100%的DNA。因此,如果总体积为35 ul,对于从5 ug质粒DNA(5 Kb)中分离的1 Kb片段而言,20 ng/ul的产量不算太差(已经回收了70%的DNA)。
我们经常发现,污染物水平或DNA制备质量,而不是DNA产量本身,常常是成功克隆的一个更重要的决定因素。我们还一直观察到,与添加稍少一点片段的相同反应相比,有时在连接中添加较多片段会降低克隆成功率。尽管这可能仅仅是因为额外的DNA末端将连接酶从载体末端带走,但添加过多的凝胶提取物可能是成因,因为制备液中来自凝胶的污染物水平低。
连接反应DNA寡核苷酸退火
寡核苷酸退火的基础原理是,两个寡核苷酸加热变性,然后通过一段时间的冷却,使两个寡核苷酸碱基配对在一起。对于定制的寡核苷酸,请参阅OLIGO)。这个过程通常用于制备短DNA片段,用于:
- 创建用于连接的shRNA DNA区域
- 创建用于连接的microRNA DNA区域
- 添加连接子,以去除或添加限制性位点。
- 需要小双链DNA区域的研究,例如DNA蛋白结合测定。
购买大量磷酸化寡核苷酸的成本可以非常高昂。由于这个原因,许多基团简单地将寡核苷酸连接到尚未去磷酸化的载体中。当要将寡核苷酸连接到已经用两个单独的具有不相容末端(防止载体自身闭合)的限制酶切割的载体中时,这特别有效。来自连接的背景可能仍然很高,但寡核苷酸通常在寡核苷酸连接中显著过量,因此反应仍然有效。
也可以首先用多核苷酸激酶(PNK)磷酸化寡核苷酸,但是寡核苷酸难以清除,因为它们很可能由于它们的长度短而不与DNA清除柱结合。然而,PNK反应在连接酶缓冲液中进行,因此这可能不是必需的,但是在寡核苷酸被置于连接之前必须将PNK热灭活,否则PNK将使载体磷酸化。
通常产生寡核苷酸的亚磷酰胺,一般可以用来产生40-50个碱基,而不会有太多错误。然而,使用该技术产生有100个碱基或更长的寡核苷酸通常会导致突变。我们经常发现,最好将长寡核苷酸分成两个较短的寡核苷酸,在相对的寡核苷酸的中间具有10-15个碱基对的重叠,以让它们连接在一起。在测序后的突变频率应该低得多。

考虑冷却步骤。我们尝试在水浴中冷却寡核苷酸,只需将其加热至95度然后将其关闭以冷却,或者在三脚架(老款)上的烧杯中将它们加热至100度,然后将烧杯放入冰水中(并且装有寡核苷酸的试管仍在烧杯中)冷却。我们还尝试将寡核苷酸置于PCR机器中,每隔30秒降5度来达到冷却目的。现实情况是,该方法并没有产生多大差异。将它们置于高温下太久会导致水解,因此我们避免仅关闭水浴。我们通常在烧杯中的水浴中将寡核苷酸加热至95度,在水浴中装有寡核苷酸的试管在eppendorf中漂浮着。然后将其置于冰水中10分钟。我们觉得这种方法似乎很有效,但也许有其他方法也适合您。
设计具有悬突的寡核苷酸可以是件困难的工作,需要重建要把寡核苷酸嵌插其中的位点。下图给出了寡核苷酸可能的样子,在这个举例中,寡核苷酸将连接NcoI和XbaI限制性位点。

DNA寡核苷酸退火实验方案
提示:寡核苷酸和引物原液通常以100 μM(100皮摩尔/ul)浓度重悬。
- 将10 μL寡核苷酸原液(假设您有两个试样要退火)和25 μL无核酸酶的水(货号:W4502)加入到1.5 ml微管中。
- 添加5 µL限制性消化缓冲液(例如100 mM NaCl(货号:S3014)、50 mM Tris-HCl(货号:93362)、10 mM MgCl2(货号:M0250)、1 mM 二硫苏糖醇(货号:D0632)),pH 7.9。缓冲液的存在有助于保持正确的pH值,从而有助于DNA的稳定性,盐应能促进退火。寡核苷酸的浓度现在为20皮摩尔/ul。
- 将烧杯中的水预热至95度,然后将试管漂浮在烧杯中5分钟。
- 将烧杯放入装有冰和水的冰盒中,使烧杯冷却10分钟。
- 烧杯中的水冷却后,将寡核苷酸在水中稀释10倍和100倍,并将1 μL稀释液加入到标准的20 μL连接反应物中。人们通常想多加一些,但这只会将连接酶从载体末端带到额外的寡核苷酸上,造成连接效率降低。连接反应中,寡核苷酸的最终浓度约为2或0.2皮摩尔/μL。这对载体而言,仍然属于寡核苷酸显著过量。
向DNA添加5‘磷酸盐
T4 DNA连接酶需要在其中一个DNA分子上连接5'磷酸盐以连接DNA,为此,在将DNA分子加入连接反应之前,通常需要磷酸化DNA分子,例如当平末端克隆PCR产物时。
DNA磷酸化实验方案
无核酸酶的水(货号:W4502):4.5 ul
PCR产物或其他DNA(已纯化):4 ul(如果您的PCR产物有5'凹陷或平末端,则将其加热至70°C 5分钟,并在冰上冷却后再加入反应中)
10 x T4 DNA 连接酶缓冲液:1 ul(使用连接酶缓冲液,因为它含有ATP)
T4多核苷酸激酶:0.5 μl0.5 ul
在37℃孵育激酶反应物30分钟。磷酸化的PCR产物无需纯化,即可直接用于连接反应(如在进行定点诱变时)。如果要将PCR产物连接到去磷酸化载体中,最好热灭活PNK酶,以防止其磷酸化骨架载体并导致高背景。这通过在65摄氏度下将反应物孵育20分钟来实现。
DNA去磷酸化
载体去磷酸化的目的是通过除去DNA连接酶将磷酸二酯DNA骨架连接在一起所需的5' 磷酸基团,在连接反应中防止其自身连接。有各种碱性磷酸酶,包括小牛肠磷酸酶(CIP)、虾和南极磷酸酶。CIP(货号:P4978)是最常见的,但难以热灭活。不同酶的温度和缓冲液可能不同,请参阅制造商的说明。
限制性消化反应中DNA去磷酸化的DNA去磷酸化实验方案
在限制性消化反应物 / 溶液中加入50-100 μl DNA(5 μg)
1-2 μl CIP酶(1单位/μl)
- 在限制性消化中加入1-2 μl CIP。CIP在大多数限制性消化缓冲液中稳定且有活性。
- 将样品在37°C孵育30-60分钟。
在TE或H2O中去磷酸化的DNA去磷酸化实验方案
将20-40 µl DNA(5 µg)加入TE(货号:T9285)或核酸酶 H2O(货号:W4502)
5 µl 10xCIP缓冲液
1-2 µl CIP酶(1单位/µl)
定容至50 µl
- 在无菌的1.5 ml eppendorf中,加入DNA,然后加入CIP缓冲液,然后加入1-2 μl CIP。
- 用移液管吸头彻底混合,在37°C孵育30-60分钟。
提示1:确保您要连接到去磷酸化载体中的片段具有5' 磷酸基团。标准寡核苷酸 / 引物和PCR产物通常不被磷酸化,必须用T4多核苷酸激酶处理(参见磷酸化5' 末端)。通常更容易将限制性位点添加到PCR产物的末端(在末端加上一些额外的碱基对),而不是磷酸化片段。
提示2:CIP储存在甘油缓冲液中以保持稳定,但这意味着它会沉到水溶液的底部。添加CIP时,将试管拿在您面前,看着CIP被滴入DNA混合物中,然后确保在孵育前将其重新正确悬浮。
DNA平末端克隆实验方案
有时需要使DNA分子的末端变平,例如:
- 制作DNA文库时
- 需要克隆到载体中的带有粗糙末端的剪切DNA
在不可能选择相容的限制性位点的情况下,需要在连接前使载体、或插入物、或者两者变钝(可能是最后的解决方案)。有两种选择,一是用DNA聚合酶(如Klenow或T4),一是用绿豆核酸酶。Klenow和T4 DNA聚合酶均填充5'悬突并嚼嗜3' 悬突。如果您需要填充5' 悬突,任何一种酶都行,但是如果您需要去除3' 悬突,则T4可能是更好的选择,因为它具有较强的3' 至5' 核酸外切酶活性。绿豆核酸酶可以嚼嗜5' 和3' 悬突。
Klenow钝化实验方案
- DNA应溶于1x限制性消化缓冲液或T4 DNA连接酶反应缓冲液中,每种补充33 μM dNTP [终浓度]。
- 每微克DNA加入1单位Klenow,在25℃下孵育15分钟。
- 添加EDTA至终浓度10mM,在75℃加热20分钟,使反应停止。
T4钝化方法
- DNA应溶于1x限制性消化缓冲液,且补充100 μM dNTP [终浓度]。
- 每微克DNA加入1单位T4 DNA聚合酶,在12℃下孵育15分钟。
- 添加EDTA至终浓度10mM,加热至75℃ 20分钟,使反应停止。
绿豆核酸酶钝化方法
- 在1X绿豆核酸酶缓冲液或限制酶缓冲液中悬浮DNA(0.1 μg/μl)。
- 每μg DNA添加1.0单位的绿豆核酸酶。
- 在30°C孵育30分钟。
请勿热灭活绿豆核酸酶,因为DNA的单链区域可能会在酶失活之前出现,导致意外降解。通过离心柱纯化,或者通过苯酚/氯仿提取和乙醇沉淀,使酶失活。
DNA连接实验方案
在克隆过程中,所有环节最终都归结于连接,因此在连接之前的所有步骤都会大幅影响连接效率。在进行连接反应之前,请务必阅读每个部分的提示和说明。
连接反应与对照反应一起进行。通常有两种对照可用:
- 只有载体,没有连接酶(未切割载体对照)
- 带连接酶的载体(当与对照1一起使用时,是去磷酸化不足的对照)
- 真正的连接,即载体 + 片段 + 连接酶。
如果对照1上有菌落,说明载体的限制性消化不起作用,因为即使没有连接酶仍得到了菌落。如果在对照2上有菌落,对照1上没有,说明碱性磷酸酶处理不起作用。这是因为虽然消化液工作良好(对照1没有菌落),但连接酶能够使质粒重新环化,因为在去磷酸化过程中没有去除5' 磷酸盐。如果在3上有菌落,在1和2上没有(或者较少),您应该祝贺自己一下,取出一些菌落,可以回家喝杯茶庆祝了。
反应条件可因实验室而异。在16°C下过夜可达最佳结果,但这意味着整个实验过程增加额外的一天(克隆周期延长为4天)。在室温下进行1-2小时的连接反应,通常会产生良好的结果,并且将完整的克隆周期缩短至3天。但头一天太长。
两种最困难的连接类型是连接PCR产物和平末端连接。与从一个载体到另一个载体的标准片段克隆相比,这些连接的效率较低。
典型的反应可以设置如下:
- 通过添加所有反应共用的组分开始建立反应,即将水加入缓冲液,然后将载体加入每个试管中。
- 然后将片段添加到连接试管中。
- 最后,将连接酶加入连接试管和正确的对照试管中。请勿涡旋反应物,因为DNA连接酶对剪切力敏感。可改用添加连接酶时所使用的吸头充分搅拌,并/或者用手指反复轻弹试管。
- 在室温下孵育1-2小时,或在16°C孵育过夜。有人在65°C下热灭活连接20分钟,但这并无绝对必要。
- 连接完成后,使用质粒转化细菌,从而直接表达蛋白质,或者培养许多质粒DNA的拷贝以供进一步使用。
提示1:连接酶缓冲液中的BSA可在冻融时沉淀出来,在缓冲液底部可见白色沉淀。涡旋,并用手握住或置于37°C下使其暂时升温,以使其重悬。
提示2:连接酶缓冲液含有ATP,在多次冻融循环后会降解。将缓冲液原液分成小等分试样,如果冻融超过3次请丢弃。
提示3:加热连接物(加酶前)至37°C几分钟以打开粘性末端,或在使用单一限制性位点连接时,帮助线性化瞬时重新环化的载体。在添加酶之前,将连接物冷却回室温,以免添加时损坏连接酶。我们并未亲试,但听说有效。
提示4:据报道,在连接前用蛋白酶K处理PCR产物有助于连接。凝胶提取PCR产物则没必要这样做,因为会用PCR清理试剂盒来纯化它。
提示5:从凝胶中提取DNA时,避免将DNA暴露于紫外线下。在某些情况下,这会导致连接效率大幅下降。
提示6:避免使用体积超过20-30%总连接体积(通常总体积为20 μl)的凝胶纯化材料。否则反应中可能含有过多的盐和其他污染物,这会降低连接效率。
提示7:如果要在琼脂糖凝胶上运行,则几乎看不到载体的量,因此大约为20-30纳克。片段应该更丰富,但其浓度不会超过载体浓度的5-10倍。载体与片段比例我们通常用1:2或1:3。
计算插入物与载体的比例
插入物与载体的摩尔比可对连接反应以及随后的转化步骤的结果具有显著影响。插入片段与载体的摩尔比可以从1:1至10:1不等,为了获得最佳结果,可能需要同时尝试多个比例。以下计算方法将告诉您片段与载体的比例为6:1(片段:载体)。将3改为任何其他数可以算出其他浓度。


请注意,只有正向引物含有突变,因此,通过保持逆向引物相同但使用不同正向引物,即可轻松地产生一系列不同的突变。上面的例子是一个3 bp替换,但插入可以相同的方式进行。如果需要非常大量的替换或插入,则可以在两个引物的5'末端引入突变碱基。
可以通过在模板上分开正向和逆向引物,来删除任何大小的片段。
由于它们的合成方式,PCR引物通常在其5'末端没有磷酸盐基团。这意味着PCR产物的末端不能简单地连接在一起,它们必须首先被磷酸化。对此有两种主要方法:1) 订购含有已添加到5'末端的磷酸盐的引物,或者 2) 用多核苷酸激酶(PNK)磷酸化PCR产物。如果您只做几个SDM并且在冰箱中没有任何PNK,使用磷酸化引物是个好办法(再加上这样做会少一步操作)。如果您要做很多SDM(可能超过10个),则使用PNK更经济有效。
第 2 步:PCR
使用校对聚合酶以避免引入任何其他突变是很重要的。也就是说,如果您仍然担心引入突变,您可以在诱变后,将突变的片段亚克隆回到相同的骨架中。
由于该方法基于PCR,因此在较小的质粒上往往效果更好。最好是遵循聚合酶附带的说明,但可以建立一个类似于下列例子的PCR:
35.5 μL水
5 μL 10x聚合酶缓冲液
1.5 μL正向引物(0.3μM终浓度)
1.5 μL反向引物(0.3 μM终浓度)
5ul dNTP(200 μM终浓度)
1μL模板 DNA(微量制剂的1:100稀释液)
0.5 μL 聚合酶
将反应物置于冰上,并在混合反应物后将试管直接转移至预热的PCR仪中。
PCR程序:
- 98°C 60秒
- 98°C 8秒
- 55-65°C 20秒
- 72°C(时间长度取决于质粒大小和所用的聚合酶类型)下多重复 / 循环步骤2-4 27-30次
572°C 5分钟
保持在室温下。
第 3 步:纯化PCR产物
在琼脂糖凝胶上进行整个反应。切除条带并使用凝胶提取试剂盒清洁DNA,用30 μl洗脱。
第 4 步:使5'末端磷酸化
* 如果在PCR中使用磷酸化寡核苷酸,则可以省略该步骤。
4.5 μl 无核酸酶的水(货号:W4502)
4.5 μl PCR产物
1μl 10x T4 DNA连接酶缓冲液*
0.5μL T4 多核苷酸激酶
在37°C孵育40分钟。
* 使用连接酶缓冲液,因为它已包含ATP,并且PNK在其中有活性。
第 5 步:连接DNA末端
将连接反应物置于冰上。
6.7 μl 无核酸酶的水(货号:W4502)
2 μl PNK反应物(来自步骤5)
0.8 μl 10x T4 DNA连接酶缓冲液
0.5 μl T4 DNA连接酶
在16°C孵育一夜,或在室温下孵育2小时。
第 6 步:转化进感受态大肠杆菌
第 7 步:通过DNA测序确认突变

如要继续阅读,请登录或创建帐户。
暂无帐户?