RT-PCR / RT-qPCR故障排除指南
制定PCR或RT-PCR故障排除方案
错误潜在来源和/或操作员人为错误问题
操作员出错的可能性很多。通常无法确认这些错误的原因。所有故障排除流程的第一步是检查实验方案,然后重做实验。检查实验方案(参见附录A,本指南的实验方案),甚至要求同事(最好是经验丰富的分子生物学家)审查实验计划,是相当重要的做法。一位博士后研究员在运行PCR多次失败后,意识到PCR预混试剂缺少dNTPs,这个警示性故事提醒我们,即使是最优秀的科学家在过度工作后,也非常容易犯简单的错误。
预混试剂
反应组分预混试剂的错误或问题可能是所有样品和阳性对照中扩增彻底失败的根源。 在重做实验之前,应检查所有组分以及浓度。如果正要使用新的一批试剂,那么在开始进行一系列重大实验之前,应先对照运行新试剂和原试剂,这是非常有效的预防措施。
在转换预混试剂时,必须认识到有些检测对缓冲液组分 / 退火温度 (Ta) / 引物浓度特别敏感。更改其中任何一项都可能会导致不同性能。因此,在进行重大改变之前,应在选定的预混试剂和所需的所有仪器上验证所有测定。查看每种预混试剂的说明书也很重要,因为说明书给出了针对给定的酶、热启动机制和缓冲液组分优化的推荐条件。
良好的实验室操作规范要求确保为所有要混合的样品制备充足的反应预混试剂。确保将所有组分小心解冻并充分混合,且在等分至样品之前,将实验预混试剂彻底混合均匀。这对于某些2×缓冲液特别重要,例如比普通PCR缓冲液更粘稠的KiCqStart®。
寡核苷酸
如果寡核苷酸序列不正确、设计不当、在非最佳浓度或非最佳退火温度(Ta)下运行、标记不充分、或淬灭(对于探针),那么寡核苷酸可能会导致问题。在非最佳条件下运行寡核苷酸的检测,或使用不当的设计,可能会产生一些数据,但这些数据可能不能反映所研究的生物的真实情况。在收到冻干的寡核苷酸后,请务必:
- 验证序列
- 确保在使用前重悬所有DNA
- 确认溶液浓度符合预期要求
加热寡核苷酸至90 °C 5分钟,然后混合均匀,促进寡核苷酸的重悬。反复冻融也会影响寡核苷酸的性能,因此应将所有寡核苷酸以原液浓度(通常为100 μM)等分,然后保存于-20°C,若需长期保持,则保存于-80°C。
在故障排除阶段,应返回至目标序列并确认寡核苷酸序列确实存在,从而验证是否订购了正确的序列。联系寡核苷酸供应商,确保寡核苷酸质量正确。测量寡核苷酸的工作浓度,并可视化检查荧光分子以确认其被标记。在SYBR® GreenI qPCR混合试剂中测试探针检测的引物以验证扩增。考虑优化引物浓度或退火温度Ta(参见实验的优化和验证)。当第一次使用探针时,对尽可能多的可能波长收集荧光数据,以便观察通道之间的任何潜在信号泄漏,并发现标记错误。
优化不足
实验优化效果详见实验的优化和验证。当检测失败或表现不佳,但在设计或操作程序方面没有错误时,则可能需要优化实验条件。排除故障时,分别在100 nM、500 nM和900 nM最终浓度下和/或在55°C至70°C退火温度Ta之间测试引物(使用温度梯度),以确定测定是否随着进一步优化而得到改善。
RT-PCR和qPCR实验方案设计
关于实验方案的设计,请参见PCR / qPCR / dPCR实验方案设计。在对实验方案进行故障排除时,请务必验证设计。确认PCR/qPCR引物和扩增子位置与RT引物实验方案一致。例如,确保应用于oligo-dT引发后制备的cDNA的测定位于转录物的3'。确保序列信息可靠,并考虑了适当的剪接变体和SNP。
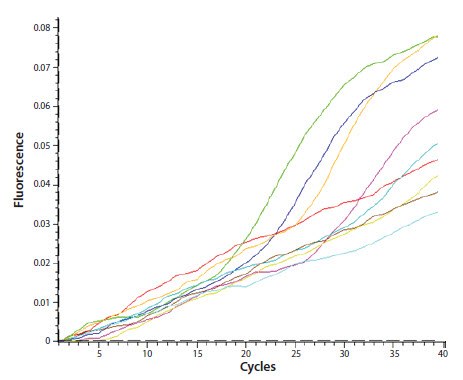
图 11.1A.测定的扩增曲线异常,具有明显的基线漂移。

图 11.1B.测定的探针序列被输入至mfold折叠预测软件中。很明显,探针可以在溶液中采用稳定的折叠结构,这很可能导致所观察到的问题。
RNA或DNA模板质量
关于模板质量对测定性能的影响,请参见样品纯化和质量评估。模板质量包括对数量的考虑、完整性和抑制剂的存在。 必须保证RNA质量符合最适当的RT引物实验方案(请参见逆转录),并尽可能使用最佳质量的模板。与此类似,添加到RT反应物中的RNA量必须在实验方案规定的范围内,且在许多情况下,对于所有反应,应该是相同的。值得注意的是,ReadyScript® 属于本指南的例外情况,因为采用该试剂及其实验方案将产生与输入RNA量成正比的线性cDNA浓度。在对产率高于预期Cq的样品进行故障排除时,请运行SPUD测定,并通过1:5或1:10连续稀释对样品进行稀释,然后重复测定(图11.2),以鉴定含有抑制剂的样品。
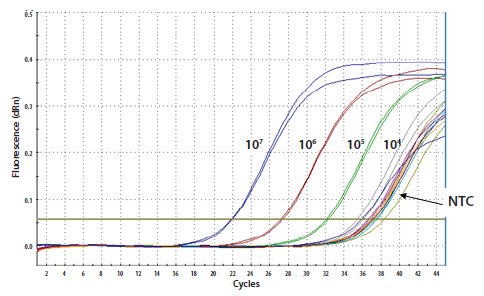

图 11.2.DNA模板10倍连续稀释液的扩增。复制是精确的,但ΔCq 值不一致,随着稀释度的增加而降低。该数据还显示无模板对照(NTC)呈阳性信号,表明污染或引物二聚体形成,且稀释到少于105个拷贝时的数据与NTC相同。
模板数量也是一个重要的考虑因素。PCR中模板太多或太少都会导致反应失败和qPCR扩增曲线看起来异常。图11.3A显示了含有10倍连续稀释的人工合成寡核苷酸模板的反应。较下部的稀释物太浓,不足以使反应有效或让仪器有效处理基线数据(图11.3B),导致扩增曲线异常和数据不可靠。
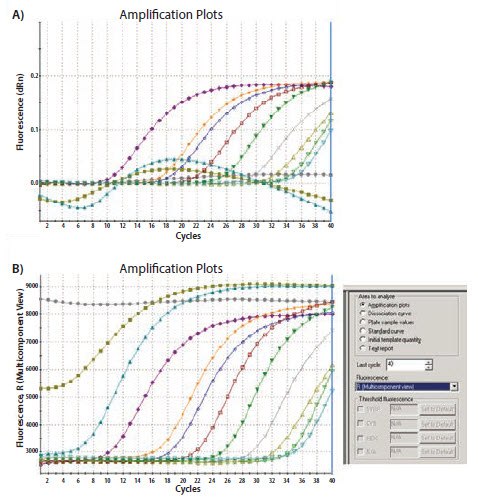
图 11.3.A)用特异性引物和FAM标记的探针扩增10倍连续稀释的人工模板。浓缩样品的Cq值极低,扩增曲线间隔不规则且异常。B)显示了这些扩增图的原始数据。含有最高浓度靶的反应也具有明显较高的背景荧光,而且在整个反应中都具有最小的荧光产率。
验证PCR程序
PCR循环条件必须适合所进行的实验和所使用的试剂(例如参见预混试剂)。不建议在未经验证的情况下采用默认仪器设置。
热循环仪故障
仪器故障的原因有时较为隐蔽,因此难以诊断。为了防止产生昂贵的维修费用,请确保所有仪器操作员均经过全面培训并在其最初操作时对其进行监督。有些仪器故障将引起严重故障,导致无扩增或荧光数据,而有些仪器故障则会使数据失真或以非均匀方式处理样品,从而在相同的生物样品中产生人为差异。使用对照样品与对照测定可有效地排除故障。当怀疑仪器出现故障时,应在所有孔中运行可靠、优化的测定。这种一致性检查将揭示仪器特定区域的问题,并区分开测定问题与仪器问题。
故障排除示例 — 诊断工具的使用
运行精心设计的PCR后,可以使用多种诊断工具来排除故障:
- 对照样品和测定
- 终点凝胶 / SYBR Green I染料试剂
- 扩增曲线(检查复制和扩增曲线图)
- 标准曲线(梯度和R2)/ 连续稀释
- 解链 / 解离曲线(SYBR Green I 染料、Molecular Beacons、Scorpions®探针)
- 原始数据 / 多组分图
对照样品 / 反应
强烈建议使用对照。如果没有适当的对照组信息,几乎无法对失败的测定进行故障排除。
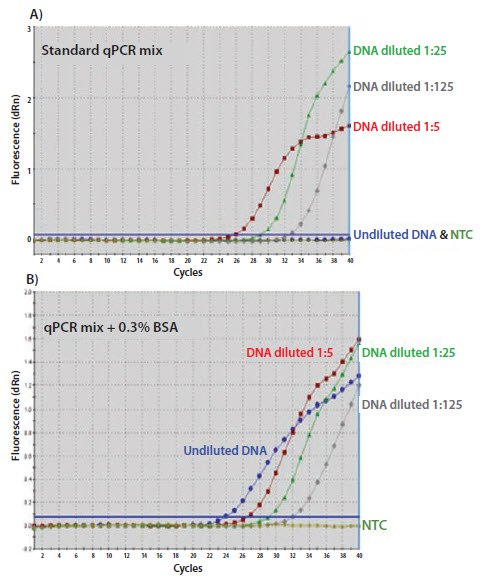
图 11.4.A)未稀释的模板扩增失败,而稀释后则显示扩增效率提高。B)向qPCR混合物添加0.3% BSA以支持未稀释模板的扩增。
由于几乎没有可用于故障排除的信息,调查完全失败的测定可能较为困难。因为许多验证失败是由于某些灾难性错误而导致的,因此首先应检查实验设置,然后重做PCR。如果失败,则故障排除流程有赖于实验的每个组成部分的信息(图11-5)。
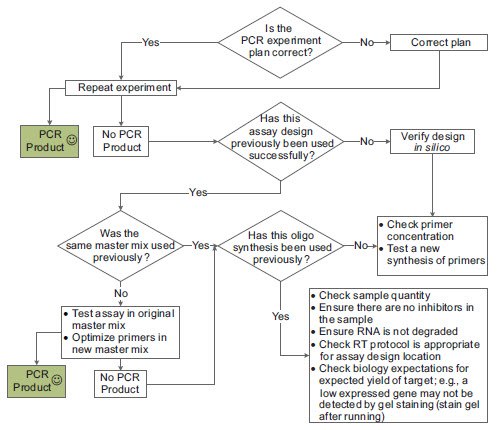
图 11.5.PCR的基本故障排除流程。
当qPCR实验完全失败时,第一步是检查来自寡核苷酸制造商的测定方案设计、寡核苷酸序列和QC数据。虽然测定可能失败,但qPCR多组分 / 原始数据可用于提供进一步的信息。图11.6A显示了含有6-FAM™或HEX™ (VIC®)标记探针的两种测定的原始数据图。尽管两种测定均显示出扩增,但HEX信号大约是FAM信号的一半。由于这是一种固有的较弱的染料,所以观察结果是正常的。琼脂糖凝胶分析(图11.6B)显示两种反应产生相似浓度的产物,进一步证实了qPCR Cq值相似的观察结果。

图 11.6.A)含有FAM和HEX标记探针的双重测定的原始数据曲线。FAM探针天然有较高的荧光产率。B)琼脂糖凝胶显示在每个反应中生成等量的产物,并证实了qPCR Cq观察结果。
检查原始数据是一个有效的检查手段,它有助于验证正确标记了探针,并将探针添加到了反应中。图11.7显示了三重实验中三个靶标扩增的原始数据。每个靶标的特异性探针用FAM、HEX和TAMRA标记。HEX和TAMRA探针显示低背景值和有效扩增,但FAM信号在整个实验中始终很高且没有扩增迹象。这一结果与反应中探针浓度过高或探针故障导致信号未初始猝灭相一致。在这种情况下,应验证探针浓度和测定设计,确保探针与标记物和猝灭剂相容,并在必要时测试新探针。
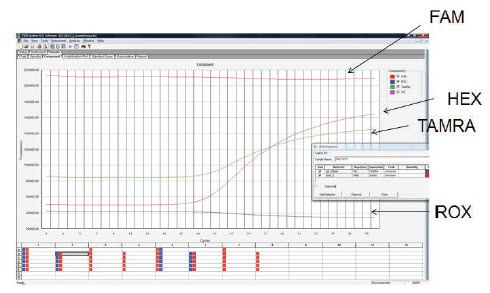
图 11.7.三重反应用于使用FAM、HEX和TAMRA标记探针检测三种靶标。HEX和TAMRA探针产生了靶标扩增,但FAM探针未显示扩增。从原始数据可以看出,背景荧光特别高,并且在整个反应中没有变化。这与反应中探针浓度过高或探针淬灭不充分造成的错误一致。
如果原始实验依赖于探针检测,则应使用SYBR Green I试剂重新测定,重新测定时要包括阳性和阴性对照(但不要用珍贵的样品)。也可在溴化乙锭染色的琼脂糖凝胶上检查失败反应的产物。首选方法是采用SYBR Green I方法重做实验,因为它可避免污染风险,并可通过重做实验来验证原来的失败。如果SYBR Green I实验产生数据,则原探针失效可能是由于技术性错误或探针故障所致。要确定是因实验错误还是因探针故障所致,请重做探针实验;如果反应再次失败,则请更换探针。可以采用这一方法来研究产生不良数据的反应。在图11.8的示例中,探针反应不佳,与使用SYBR Green I的反应相比较,可以看出探针信号不反映实验。在这种情况下,应验证测定设计并测试新探针。
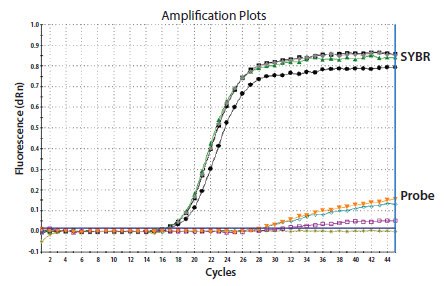
图 11.8.用含有qPCR探针或SYBR Green I染料(图中所示)运行了相同的反应。SYBR Green I反应约有11个循环更敏感,产生的终点荧光高得多。这表明探针存在故障或探针设计存在问题(数据由英国Stephen Bustin教授提供)。
验证探针标记
原始数据或多组分曲线是一种有用的诊断工具,用于检查反应中是否包含适当浓度的探针以及是否已充分标记和淬灭。图11.9显示了包含3个探针的反应的多组分曲线。前两个产生扩增曲线,且背景荧光较为明显。第三个探针没有数据,从原始数据可以看出,背景荧光与不含任何探针的水空白对照样相同。因此,该数据是因为反应中不存在荧光所致。这可能是由于在设置过程中出现错误,未包含探针或未标记探针。
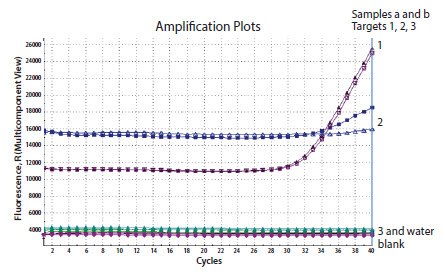
图 11.9.在同一模板样品中检测了三个基因。两个反应导致扩增(1和2),但第3个反应呈阴性。从多组分图可以看出,第3个反应的背景荧光等于水对照样,表明没有信号。
可以使用DNase I消化进一步检查探针标记。必须非常小心地进行此操作,以确保探针和引物原液不会被酶污染,否则会导致灾难性结果。把与反应中包含的试样相等的失败探针(图11.10A)等分试样(例如300 nM)在含有和不含有DNase I的情况下孵育。这可以实时进行(图11.10B),这样可以相对于时间测量荧光产率,或者在反应的起点和终点(10分钟后)读数,也能提供足够信息。进行此测试时,应将数据与功能良好且具有相同荧光标记和猝灭剂的探针进行比较(图11.10B)。
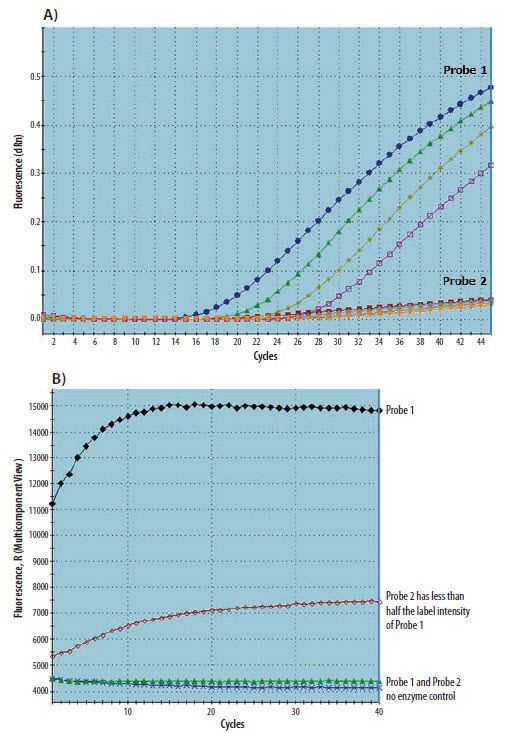
图 11.10.A)用FAM标记的不同探针检测两个模板。其中一个探针检测到的荧光信号很高,而另一个探针则弱很多。B)在存在或不存在DNase I酶的情况下,将对照和测试探针(300nM)在37℃下在实时仪器中用DNase I缓冲液孵育。探针1的荧光释放大约是探针2的两倍,这表明探针2标记不足。
扩增曲线
扩增曲线的结构和技术复制的再现性提供了关于qPCR测定质量的大量信息,还可以在第一时间发出异常报警。图11.11A中的扩增曲线是非典型的,非常凌乱,难以准确解释。对dR荧光值的进一步研究表明,终点荧光产率仅为400单位,表明反应不充分,但扩增曲线由仪器软件产生并自动缩放。类似地,图11.11B中的数据在曲线开始时呈明显的狐尾状(下降曲线),在基线部分之后再次增加。狐尾状在两个反应中是一致的,但是一个反应的终点低得多(图11.11C),导致狐尾相对放大。
图 11.11.A) 由于具有低荧光不良数据的仪器软件自动缩放而产生噪声的扩增曲线。B) 产生低端点dR的反应具有明显的初始狐尾。C) 如果与高质量测定成比例,狐尾视为正常结果。
类似地,图11.12A中的扩增曲线显然是异常的,不能像其所示那样使用。低于零dR的扩增曲线图(图11.12A)是不当基线设置的典型结果。从该反应的原始数据(图11.12B)可以看出,实际的扩增曲线正常,表明分析的数据是仪器软件问题所致。可以从原始数据中推导出正确的基线并应用于软件中。在这种情况下,循环6至16代表反应的初始线性、基线阶段,并且在应用时,将产生正常的扩增曲线(图11.12C)。
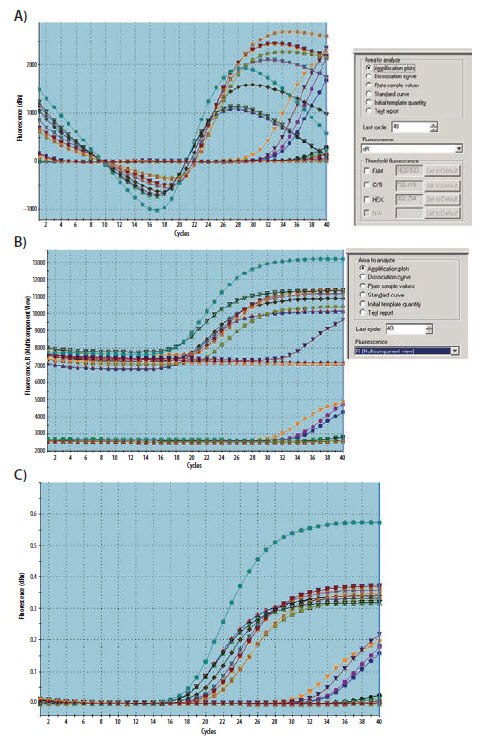
图 11.12.A)扩增曲线明显异常,其中一部分曲线下降到基线以下。B) 从原始数据曲线可以看出,反应数据与预期一致。C) 根据适当的循环设置仪器基线,根据分析的扩增曲线的数据恢复正常曲线。
扩增曲线图还可用于提供关于测定和优化质量的信息。图11.13显示了对10倍连续稀释的模板的扩增,每个浓度运行两次qPCR。两次重复运行的再现性很差,数据之间的循环差异 (ΔCq) 不是恒定的,并且不是10倍连续稀释所应有的3.323个循环。鉴于这是标准曲线,从扩增曲线可以看出,该测定未能达到标准,不能用于分析。需要进一步调查原因,但可能是因为测定设计不佳(请参见测定的优化和验证),或者不是最佳测定条件(请参见PCR / qPCR / dPCR测定设计),也可能是移液不当(重做测定)。
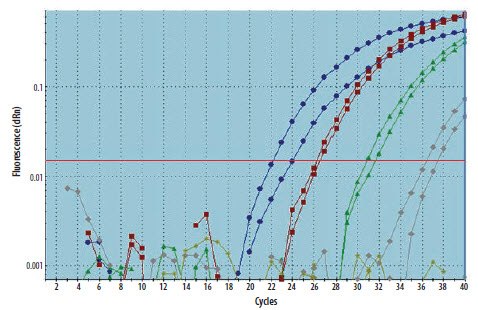
图 11.13.通过10倍连续稀释对cDNA样品加以稀释,并使用重复qPCR检测每种稀释度的特异性模板。复制结果差,表明移液或测定优化存在问题。
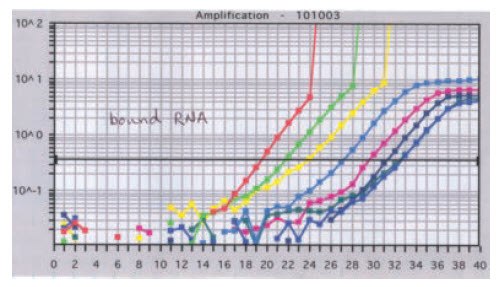
图 11.14.在标准qPCR期间,数据突然以非典型曲线上扬。
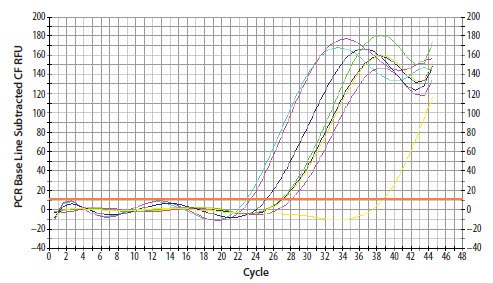
图 11.15.如果荧光曲线下降或弯曲,可能的原因是互补链与引物和/或探针竞争退火至模板。如果Ct不受影响,则请忽略。
解离/熔解曲线
解离或解链曲线分析在qPCR之后运行,是与DNA结合染料(例如SYBR Green I)或非降解探针(例如Molecular Beacons或Scorpions® 探针)结合使用的分析工具,旨在验证单个产物是否已被扩增。PCR扩增后,将所得扩增子在逐渐升高的温度下孵育,检测随着DNA从双链变为单链状态荧光信号的变化。当反应含有单个扩增子时,均匀解链,dF/dT(荧光相对于温度的变化率)曲线显示单峰。 结合来自对照的数据来检查解链曲线特别有效。图11.16A 显示了一系列实验测试样品、阳性对照和无模板对照的qPCR后解链曲线。测试样品的解链曲线与阳性对照相同,均显示dF/dT的单峰。无模板对照的解链曲线轮廓更宽,解链温度Tm更低。这些观察结果与阴性对照中引物二聚体的明显存在的情况一致。使用溴化乙锭染色的琼脂糖凝胶证实了这一点(图11.16B),图中还显示,当模板浓度低时,引物二聚体变得明显。如果在靶标浓度低的样品中检测到,这会造成过高估计靶标。因此,应优化或重新设计测定。相比之下,图11.16C表明,无模板对照中产物的解链曲线与阳性对照和测试样品的解链曲线相同。这清楚说明在实验设置期间,无模板对照被模板污染。最后一个示例显示了对cDNA样品中存在的gDNA靶标扩增的识别(图11.16D)。来自gDNA的扩增子较长,因此
解链温度Tm比来自cDNA的扩增子更高。
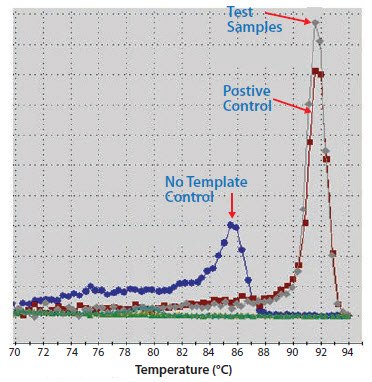
图 11.16A.阳性对照、测试反应物和无模板对照(NTC)均被扩增,然后都进行了PCR后解链分析。在无模板对照中的明显的产物的解链温度较低,解链峰更宽,这与引物二聚体形成一致。
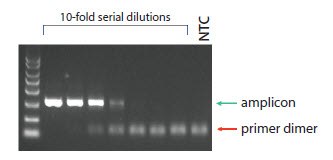
图 11.16B.引物二聚体在这些样品(及其他样品)的凝胶分辨率上是明显的,引物二聚体形成与输入模板浓度成反比。
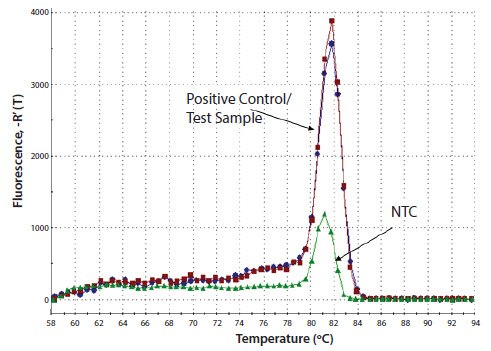
图11.16C.用解链曲线分析来鉴定NTC中反应污染的示例。
图11.16D.鉴定由gDNA PCR引起的较大扩增子。
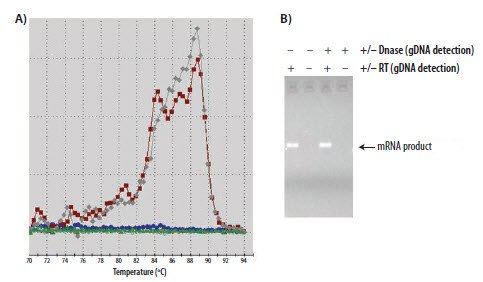
图 11.17.A)解链曲线和 B)SYBR green I反应的琼脂糖凝胶分析。虽然解链曲线显示了具有变化解链温度(Tm)的产物,但凝胶图像指出存在单一的扩增子。这表明存在含有AT或GC富集区的扩增子序列,或者导致不规则解链的重复性元素
模板的连续稀释 / 标准曲线
无论实验设计是否包括对最终定量的标准曲线的要求,检测适当模板的连续稀释是测定验证和故障排除的有效方法。检测连续稀释可以定义测定的实验线性动态范围。图11.18A显示了一个标准曲线,其低浓度数据点没在线性曲线上。这种数据模式的最常见原因是引物二聚体已在低浓度样品中形成(如图11.18B所示)。这是从图11.2中显示的数据生成的标准曲线。图11.18C显示了高浓度样品偏离线性范围的标准曲线。这种情况最常见的原因是反应中的模板抑制或基线设置不当。。
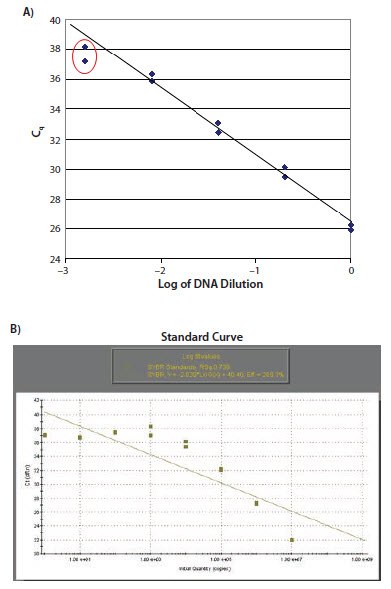
图 11.18.A)与较低浓度的靶标有关的数据点不在标准曲线上。B)这是产生如图所示的引物二聚体的典型反应。在这种情况下,对于低浓度样品,未观察到Cq的增加。
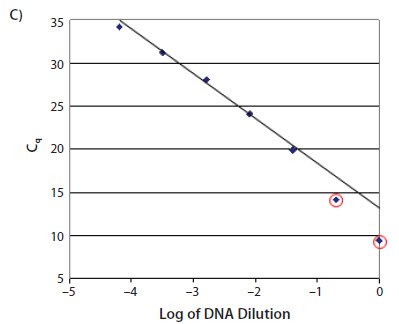
图 11.18C.具有高浓度模板的样品不在标准曲线上。这是因模板浓度抑制或基线设置不正确而产生的典型反应。
标准曲线还用于测量在动态稀释范围内的反应效率。必须注意确保用于效率计算的所有点都在线上。反应必须尽可能接近100%的效率,对那些具有明显高(> 110%)或低(<85%)效率的反应应进行进一步的调查。
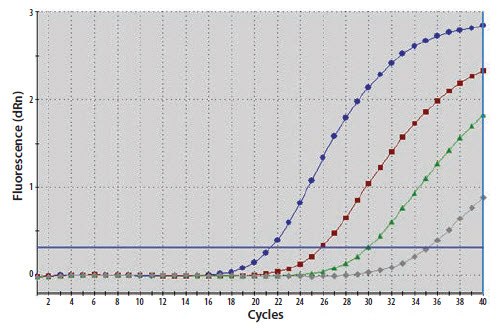
图 11.19A.模板核酸通过10倍连续稀释加以稀释。扩增曲线具有异常浅的梯度,ΔCq是4个循环而不是预期的3.3个。
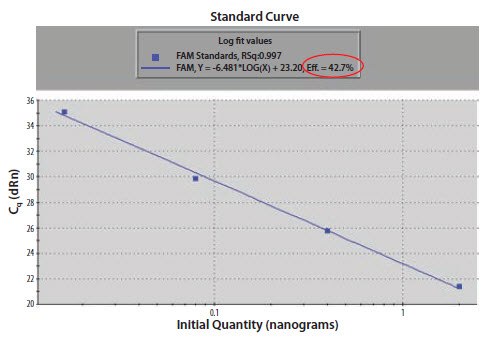
图 11.19B.Cq与数量的标准曲线梯度用于计算反应效率。
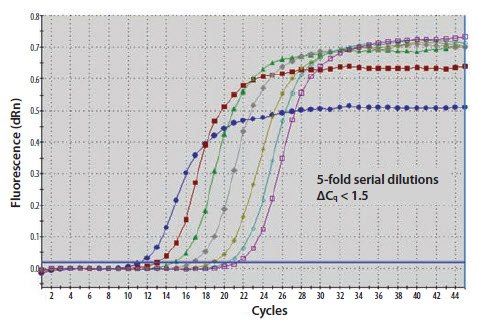
图 11.20A.模板核酸通过10倍连续稀释加以稀释。扩增曲线之间的ΔCq是1.5个循环而不是3.3个循环。
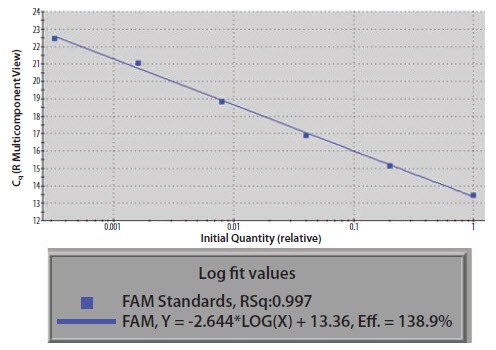
图 11.20B.Cq与数量的标准曲线梯度用于计算反应效率,反应效率接近140%。
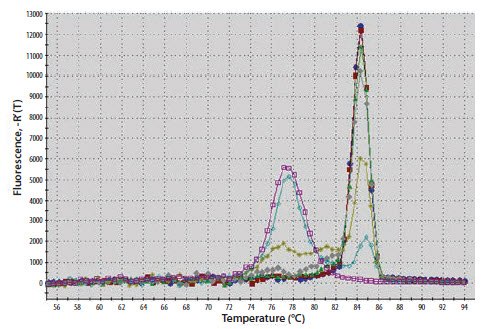
图 11.20C.从解链曲线图可以看出,较低浓度的样品(黄色和蓝色曲线)还含有来自扩增的引物二聚体的信号(在较低的解链温度Tm时达到峰值)。
RT-PCR故障排除案例研究
探针检测失败
基于探针的测定用来检测人cDNA样品中的EIFB1,但未显示扩增。使用相容试剂在ABi StepOne仪器上进行初始反应。尝试使用200nM至900nM浓度范围优化引物(图11.21),但未得到改善。测定设计已经过验证,发现其适合于靶标,而且,在硅胶中,预计是高质量的测定。 合成了新引物,并使用不同的操作员、SYBR Green I试剂(因此采用的是不同的试剂)和仪器(Eppendorf Realplex)与原始合成试剂的等分试样一起运行(图11.22)。采用这种方法时,我们知道主要目标是解决问题,次要目标是解释失败原因。该反应从两批引物产生了相同的扩增。在此阶段,反应问题似乎是探针引起的,因此合成了一个新探针,由第二个操作员在Realplex仪器上使用来自Sigma的LuminoCt® 试剂(与最初试用的试剂不同)比较了两个批次(图11.23) 。两个探针均产生扩增数据,新探针看起来略好于原探针,但值得注意的是原探针已用于测试实验室,因此已在溶液中被置于室温下数天。在此阶段,由第二个操作员在Realplex仪器采用LuminoCt® 试剂运行时,显然原测定和替代测定均起作用。
因此,最初失败的其余原因可以考虑是:
- 操作员:实验由经验丰富的科学家重复进行了多次,因此,不太可能是因为这个原因。
- 仪器:由于其他一些验证失败,所以可能有一些问题。
- 试剂:最容易测试的解释。第一个操作员在ABi StepOne仪器上使用两个寡核苷酸批次,将LuminoCt® 试剂与现有试剂进行了比较。用原试剂反应失败,但用LuminoCt®则表现出良好的扩增(图11.24)。
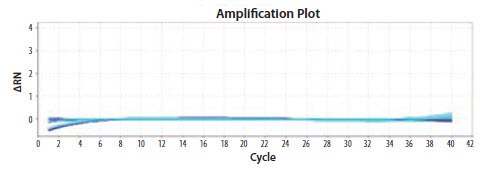
图 11.21.在200nM至900nM的浓度之间测试了EIFB1引物。在任何条件(Sigma寡核苷酸和ABi StepOne Plus上的ABi试剂)下均未观察到扩增。
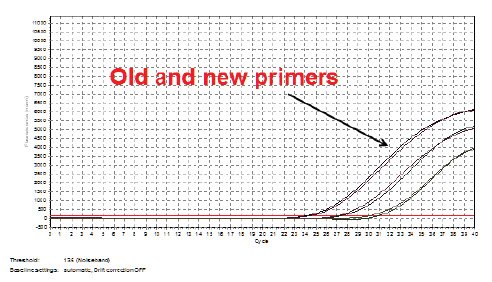
图 11.22.在SYBR Green I试剂中比较了两批EIFB1引物:原始失败的批次和新批次。(Eppendorf Realplex仪器上使用了寡核苷酸和试剂)。两组引物均支持扩增。
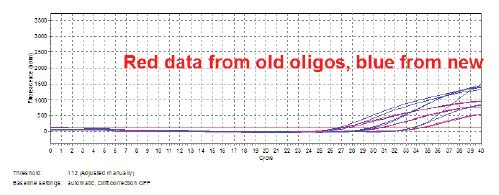
图 11.23.在Sigma LuminoCt试剂中比较了两批EIFB1引物和探针:原始失败的批次和新批次。(Eppendorf Realplex仪器上使用了寡核苷酸和试剂)。两组寡核苷酸均支持扩增。
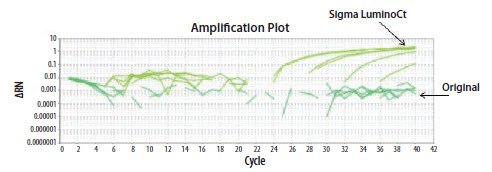
图 11.24.在两种不同的试剂(Original ABi或Sigma LuminoCt)中运行了EIFB1引物和探针测定。仅在用Sigma LuminoCt试剂的测定中获得了数据。
反应效率不正确且可变
使用标准引物和探针测定对人工寡核苷酸的连续稀释液进行测试。该测定最初是在另一台不同的仪器上开发和优化的,但是当转移到不同的测试实验室和仪器时,意外出现了异常的稀释效应(图11.25A)。所有测定条件都经过重新优化,以适应新实验室,但数据未发生变化。操作员观察到,使用相同的连续稀释液在几小时内重复测定时影响更明显。作为故障排除过程的一部分,由另一名操作员在另一台仪器上测定,该操作员再次生成预期的标准曲线。这让我们认定最初的问题是因为:操作员错误、仪器故障或实验程序中的一些细微变化。由于两位操作人员经验都很丰富且仪器在其他实验中运行良好,因此检查了细微差异。一个重要的线索是在4℃下储存样品一段时间后,发现来自相同连续稀释的数据发生了变化(图11.25A和11.25B)。因此,检查了用于连续稀释的试管并对替代品进行了测试。在换成用于连续稀释的Eppendorf 1.5 mL反应管后,生成了所预期的标准曲线(图11.25C),这表明选择适用于分子生物学的低吸附塑料器皿用于PCR,并认识到这些测定对实验方案的细微变化非常敏感,这是十分重要的。
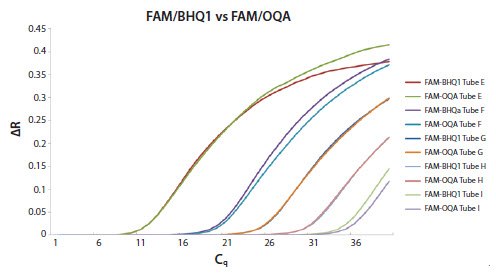
图 11.25A.将人工寡核苷酸模板稀释了10倍,并使用基于特异性探针的测定法检测。扩增曲线之间存在不一致的差异。
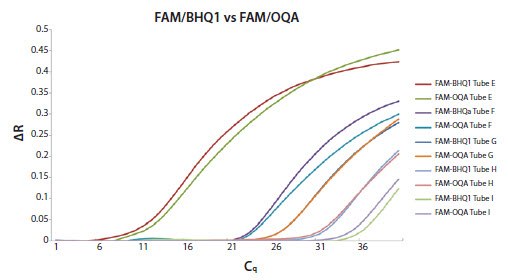
图 11.25B.将人工寡核苷酸模板稀释10倍(这些是图11.25A中检测的稀释液),并在使用基于特异性探针的测定法检测之前,在4℃下静置数小时。扩增曲线之间存在不一致的差异,这些差异随稀释与测试之间的时间延长而加剧。
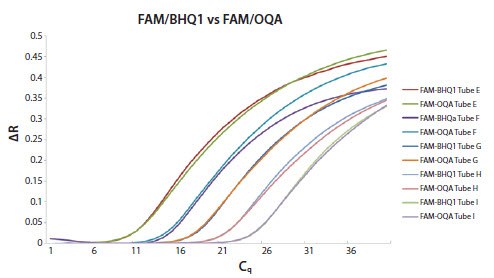
图 11.25C.将人工寡核苷酸模板稀释10倍移至分子生物学级试管(Eppendorf)中,然后使用基于特异性探针的测定法检测。正如预期的那样,扩增曲线之间存在一致的差异。
如要继续阅读,请登录或创建帐户。
暂无帐户?