阿达木单抗(adalimumab)肽图谱 LC-MS/MS 分析工作流程
肽图谱工作流程
现已开发出一种工作流程,可减少单克隆抗体酶解过程中的伪影形成。此工作流程具有以下特征:
- 蛋白质样品变性、还原、烷基化和消化分步说明
- 过滤辅助样品制备 (FASP) 完整产品列表和详情
- 低伪影酶解缓冲液 (LADB),可最大限度地减少脱酰胺和氧化
- 色谱分离与质谱检测
1.简介
充分严谨地鉴定治疗性单克隆抗体 (mAb),对于确保产品安全性和有效性至关重要。通过确定每种治疗性蛋白质的许多关键质量特性 (CQA),然后证明每批材料都在可接受的限度范围内,可确保这一目标。这一策略适用于创新疗法以及正在考虑批准的生物仿制药。在本应用说明中,我们对创新药 Humira(阿达木单抗)进行了分析。
肽图谱是一种自下而上的蛋白质鉴定方法,可以分析蛋白质的一级结构及其翻译后修饰 (PTM),如氧化、脱酰胺和糖基化。基于高分辨率质谱的鉴定方法通常称为多属性方法 (MAM)1,其中的肽图谱可通过单次分析获取蛋白质的多重信息。此类方法可获取定性和定量信息,对于全面、准确鉴定治疗性蛋白质非常重要。此外,在样品制备过程中最大限度减少蛋白质降解(例如氨基酸氧化和脱酰胺)对于准确鉴定治疗性蛋白质也至关重要。2
本文以阿达木单抗为例展示了优化的胰蛋白酶酶解和肽图谱方法。阿达木单抗是一种重组人 IgG1 mAb 糖蛋白,含有 1330 个氨基酸,靶向人肿瘤坏死因子 (TNF) 。
该方法使用过滤辅助样品制备 (FASP)3方法,以及可最大限度减少氨基酸氧化和脱酰胺作用的专有缓冲溶液。FASP 方法的优势在于,免去了去垢剂或试剂去除所需的多轮洗涤。简而言之,蛋白质在小截留分子量 (MWCO) 柱中酶解之前会经过变性、还原和烷基化。
变性使蛋白质从天然状态展开,更易酶解。使用三(2-羧乙基)膦盐酸盐 (TCEP) 进行的还原步骤可破坏连接蛋白质不同片段的二硫键,进一步暴露蛋白质。通过烷基化步骤,可让碘乙酰胺 (IAM) 与游离硫醇基团反应,防止二硫键重新形成。通过高度特异性的蛋白酶胰蛋白酶将蛋白质切割成适当大小的肽段——也可使用多种蛋白酶的组合来获取更完整的 PTM 位点信息。酶解后,通过 RP-LC-MS.M 分离肽和糖肽并分析。
为确定 RPLC-MS 分析仪器的适用性,在每次运行前对 14 种同位素标记肽的混合物进行分析,进行系统适用性测试。
某些情况下,可能需要在酶解后定量样品中的特定 mAb。为此,可采用同位素标记的阿达木单抗以及其他几种单克隆抗体作为定量内标。使用此类内标的主要优点是通过重质量标签全程跟踪和核算样品制备和分析过程的每一步。
2.过滤辅助蛋白质酶解一般步骤
在 30 kDa 截留分子量过滤柱中进行样品制备——简化了试剂清洗和去除步骤。在酶解步骤完成前,滤膜截留目标 mAb,随后肽可通过滤膜,同时去除胰蛋白酶。下图为样品制备步骤示意图。
2.1 试剂制备
~8 M 尿素溶液
取 2.25 g 尿素溶解在 3 mL 水中。混合时可稍稍加热溶液助溶,但温度不得高于 30 °C。 加热至 30 °C 以上会促进异氰酸酯形成。始终使用新鲜制备的尿素溶液,避免异氰酸酯形成及蛋白质氨可能甲酰化。
100 mM TCEP 溶液
取 200 μL 0.5 M TCEP 溶液加入 800 μL 8 M 尿素,配制 100 mM TCEP。
100 mM 碘乙酰胺 (IAM)
取 1 瓶溶解在 606 μL 8 M 尿素中,获得 500 mM 原液。将 200 μL 500 mM IAM 原液与 800 μL 8 M 尿素混合,进一步稀释为 100 mM 工作溶液。IAM 在溶液中酶解很快,因此请务必使用新鲜制备的溶液并丢弃未用材料。
SOLu-Trypsin蛋白酶
该产品以 1 mg/mL 即用浓度提供,并可在 2-8 °C 下保存长达两年。
低伪影消化缓冲液 (LADB)
该产品可直接使用,并可在 2-8 °C 下保存长达两年。MSRT1
14 种同位素标记肽的混合物。有关系统适用性准备说明,请参阅产品数据说明书。
2.2 样品
阿达木单抗标准样品 (Humira) 来自Abbvie, Inc. (North Chicago, Il),并使用以下方案进行分析。
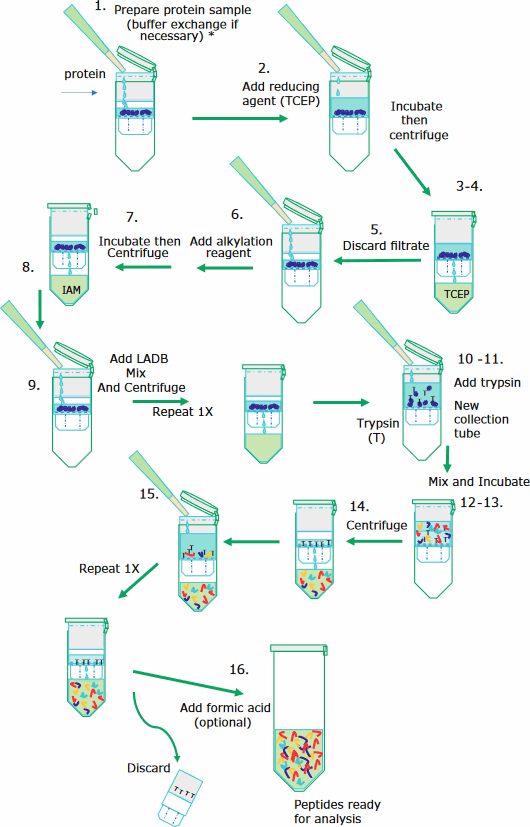
分步说明:如下图所示
mAb 按下述方式变性、还原、烷基化和酶解:
- 最好起始材料为 50 μg 以上蛋白质。样品溶液不应超过100 μL。如果体积小于 20 μL,则加水补满 20 μL。
注:如果确知样品溶液干扰还原或烷基化,请将缓冲液更换为 8 M 尿素。 - 加 100 μL 100 mM TCEP(pH 6.5)。37°C 下 300 rpm 混合孵育 30 分钟。
- 将还原后的样品转移至收集管中的 30 kDa Microcon® 过滤器中。
注:请勿使用 Amicon® 过滤器或除 Microcon 硝化纤维素 30K 截止滤膜以外的任何类型滤膜。 - 14,000 x g离心5分钟。
- 丢弃收集管中的流通液
- 加 100 μL 100 mM IAM 并涡旋 1 分钟。在环境温度的黑暗环境下混合孵育 20 分钟。
- 14,000 x g离心5分钟。
- 丢弃收集管中的流通液。
- 加 150 μL LADB 并 14,000 x g 离心 5 分钟*。再次重复此步骤。
- 将过滤装置转移至新的收集管中。每过滤装置加 100 μL LADB。
- 加入足量的 1 mg/mL SOLu-Trypsin蛋白酶,使酶与蛋白质的比例约为 1:10,涡旋 1 分钟。
- 用封口膜封住过滤装置以防蒸发。
- 将过滤装置在 37°C 下 300 rpm 混合孵育 4 小时。
- 将过滤装置 14,000 x g 离心 5 分钟*
- 加 40 μL LADB 缓冲液并 14,000 x g 离心过滤装置 3 分钟*。再次重复此步骤。
*注:如果尚未完全收集溶液,请再离心 2 分钟。离心时间请勿超过必需时间;离心时间过长往往会加剧氧化。 - 取 2 μL 纯甲酸加入滤液,涡旋并短暂离心。(此为可选步骤,因为某些肽可能在低 pH 下沉淀)。
更高序列覆盖度须知:
- 如果最初可用的蛋白质少于 50 μg,则可以使用真空干燥。这可进一步浓缩样品。
- 部分较大的疏水性肽可能无法通过 FASP 滤膜。为了防止这些肽损失并在胰蛋白酶酶解后获得更高的序列覆盖度,请将滤膜上方的溶液放入一支新收集管中。14,000 x g 离心过滤器装置 5 分钟。加 40 μL LADB,再 14,000 x g 离心过滤器装置 3 分钟。再次重复此步骤。合并所有溶液(过滤和未过滤的)。
2.4 仪器参数
LC-MS 系统参数
2.5 数据分析
原始 MS 文件经过 BioPharma Finder™ 3.0 (Thermo Fisher Scientific) 进行肽图谱分析。通过针对阿达木单抗序列搜索处理后的数据,进行肽鉴定。使用的搜索参数如下:选择中等特异性的胰蛋白酶作为蛋白酶,羧甲基化(+58.005 Da)设置为Cys残基静态修饰,Met和Trp氧化(+15.995 Da),Asn和Gln脱酰胺(+0.984 Da),Glu焦谷氨酰胺化( N端Glu的-18.011 Da)、残留的C端Lys (+128.095 Da)以及常见的单克隆抗体糖基化(如G0、G0F、G1F、G2F)设置为可变修饰。筛选数据,仅报告质量公差为 ±15 ppm 的肽。
3.结果
3.1 系统适用性
对于分析的每批样品,最好通过系统适用性测试评估仪器性能(图 1)。MSRT1 是 14 种合成肽直接进样标准品,用于评估 LC-MS 系统性能、比较 LC 梯度和色谱柱或监测其他系统变化。MSRT1 还用于预测跨 LC-MS 平台的肽保留时间。4每个肽都包含重同位素标记的氨基酸,以避免干扰样品中的肽。MSRT1 可用于多反应监测 (MRM) 实验,建议的离子对列表请见www.sigmaaldrich.com的产品页面。
图 1显示了 MSRT1 的色谱图,以及这组肽在所用色谱条件下获得的分离效果。许多实验室会在样品组进样之前、进样期间和完成进样时进样此类标准品,评估系统性能。
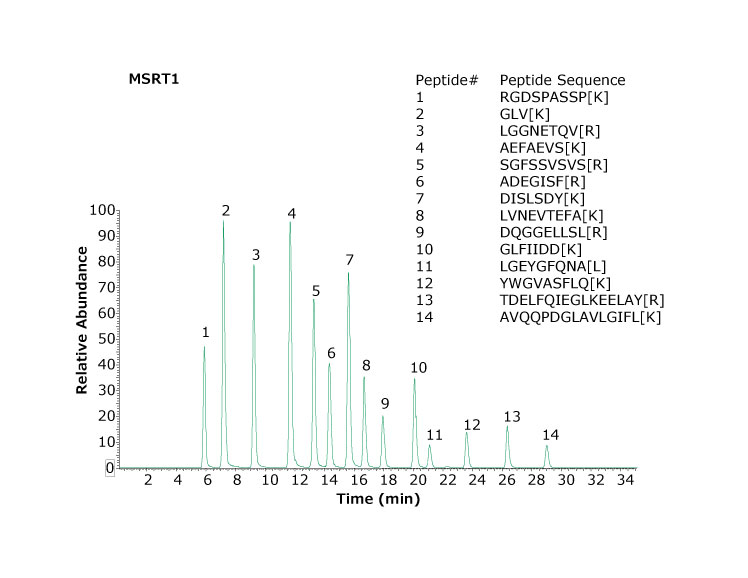
图 1.使用两根 Ascentis™ Express Peptide ES-C18 色谱柱(1.0 mm x 150 mm,2.7 μm 粒径)分离的 MSRT1 肽基峰色谱图。插图中括号内的氨基酸表示同位素标记氨基酸。
3.2 肽图谱
传统的 LC-MS 肽图谱分析程序通常很麻烦,需要在更高的 pH 和温度下对样品进行过夜蛋白酶酶解。在这些步骤中可能发生天冬酰胺脱酰胺和蛋氨酸氧化,具体程度取决于不同的条件,如所使用的试剂、离子强度、温度、pH、孵育时间、消化缓冲液以及是否存在痕量金属(蛋氨酸) 5此处提供的方法旨在最大限度减少伪影形成,从而更准确地测定内源性脱酰胺和氧化水平。此外,Microcon® MWCO 过滤器的运用简化了整个过程。
图 2显示了使用所述色谱柱和仪器条件获得的色谱图(包括高度亲水性肽的鉴定)。将两根色谱柱连接在一起可稍稍改善亲水性肽截留,由于 Fused-Core® 颗粒可实现更低的背压,这一目标完全可能实现。24.03 分钟洗脱的糖肽产生的谱图和结果如图 3所示。
图4显示了阿达木单抗获得的序列覆盖度图,表1总结了序列覆盖度结果。通过端到端连接两个 15 cm Ascentis® Express Peptide ES-C18 色谱柱实现优势,再次证明阿达木单抗重链和轻链覆盖度均超过 93%。
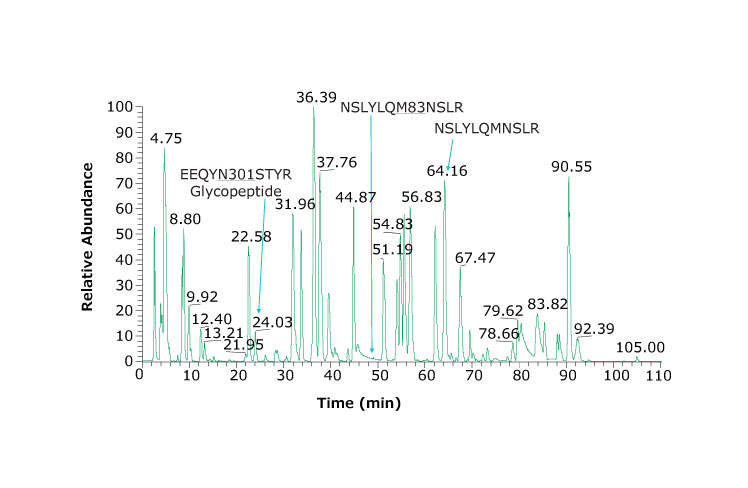
图 2.阿达木单抗胰蛋白酶肽的基峰色谱图。在 24.03 分钟时观察到重链糖基化肽 (EEQYN301STYR)。我们使用双柱设置,观察到 PGK、SCDK、VDK 等亲水性肽分别在 3.95、2.57 和 2.76 分钟洗脱。示例肽及其氧化形式是 NSLYLQM83NSLR 和 NSLYLQMNSLR,分别在 49.30 和 64.16 分钟洗脱。
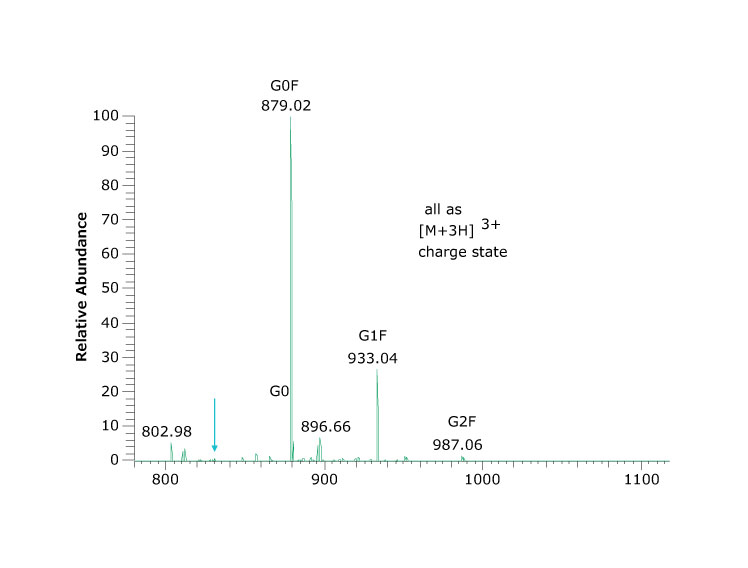
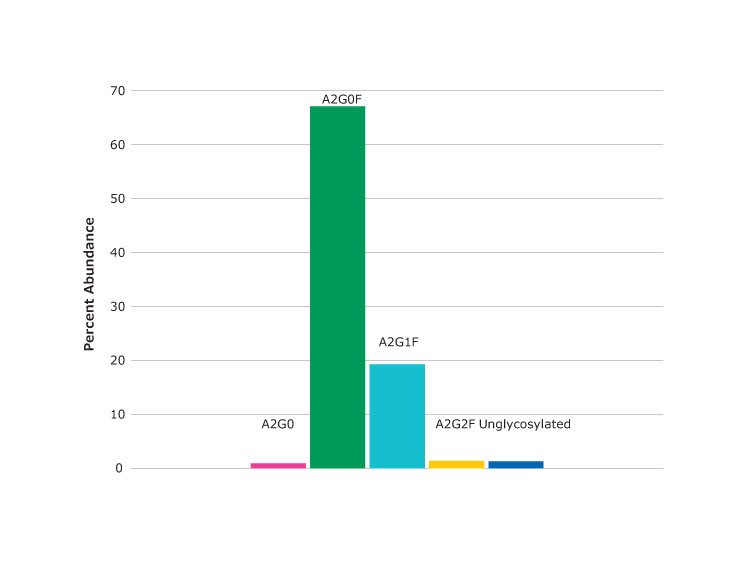
图 3.天冬酰胺 N301 处发现的聚糖光谱(上图)和丰度百分比(下图),肽聚糖形式(如图 2 所示)在 24.03 分钟处出现峰值。

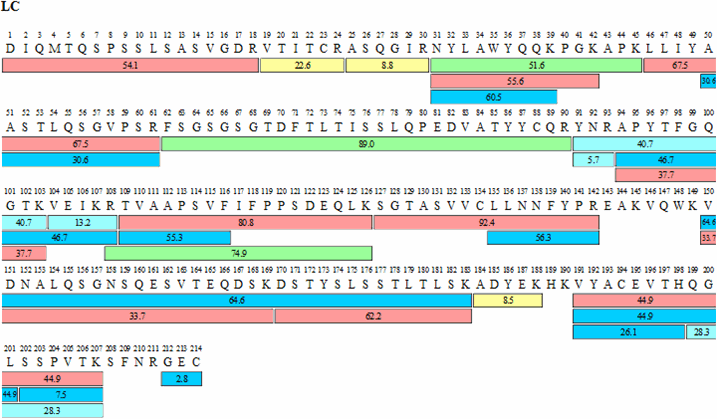
图 4.通过 FASP 胰蛋白酶酶解和上述色谱程序获得阿达木单抗重链 (HC) 和轻链 (LC) 序列图。每个条中的数字表示每个肽的保留时间,而颜色随着图示方案的信号强度变化。
上述低伪影消化缓冲液和操作过程有助于最大限度减少样品制备阶段可能发生的氧化和脱酰胺。在此操作过程中,基本上未观察到脱酰胺作用(小于1%)。如图 5所示,多个蛋氨酸位置发现了氧化,但氧化程度低于 5%。图 6显示了含有 M83 氧化位点的肽(已标记)。图 6中的色谱图显示了缺失切割实例,尽管并未达到显著程度。
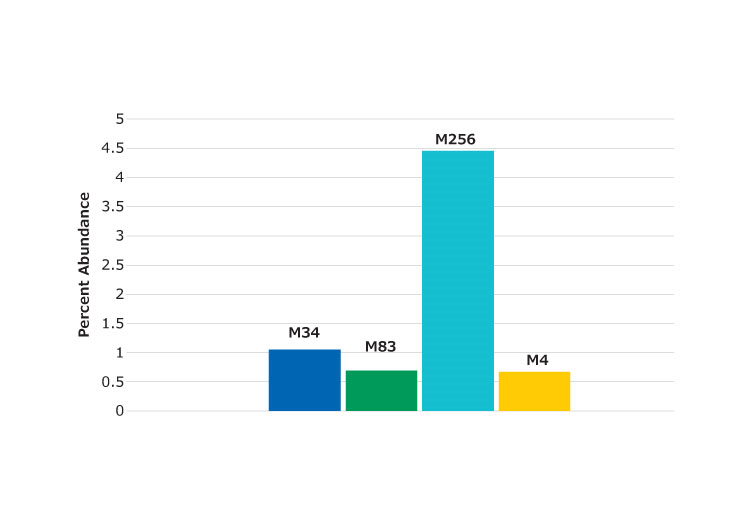
图 5.发现的氧化肽相对于未氧化形式的百分比。标签表示蛋白质中蛋氨酸残基的位置。
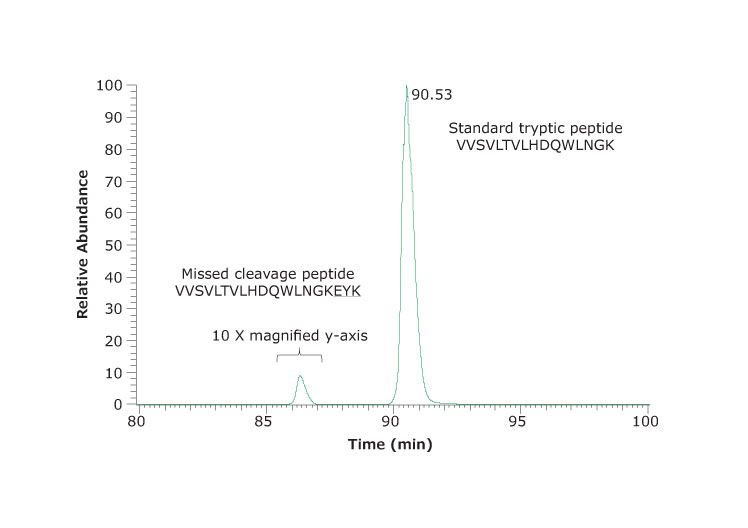
图 6.缺失切割位点示例。VVSVLTVLHDQWLNGK 和相应肽 VVSVLTVLHDQWLNGKEYK 的提取离子色谱图,其中有一个缺失切割位点。缺失切割肽占总数的0.6%。鉴定出的另一缺失切割肽丰度不明显。
4.结论
我们针对单克隆抗体治疗制剂的肽图谱分析,开发出了完整的 LC-MS/MS 工作流程。此工作流程具有以下特征:
- 使用 14 种同位素标记肽的合成混合物进行系统适用性测试
- 用于单克隆抗体变性、还原、烷基化和酶解的过滤辅助样品制备分步操作流程
- 使用低伪影酶解缓冲液进行 4 小时快速酶解
- 高序列覆盖度
- 包含所有试剂、耗材、色谱柱和相关产品的完整列表
所用低伪影酶解缓冲液和操作流程有助于最大限度减少氨基酸脱酰胺和氧化,从而更准确地鉴定蛋白质产品、理解上下游工艺。
阿达木单抗酶解在 4 小时内完成,序列覆盖度超过 93%。酶解缓冲液可最大限度减少酶解步骤中的脱酰胺和氧化,同时维持良好的酶解效率。缓冲液 pH 针对酶解需要进行了优化,并含有专有的抗氧化剂。
色谱运行期间几种聚糖型肽的测定结果也证明了肽图谱的多属性性能。
SOLu-Trypsin蛋白酶是一种专有的重组胰蛋白酶液体制剂(在毕赤酵母中表达的猪序列),以溶液形式稳定低温储存。FASP 酶解方案简化了缓冲液置换、洗涤和样品制备任务。
相关产品
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?