Universal SYBR Green qPCR Protocol
Technology Overview
Assay Considerations
Methods of Quantification
Equipment & Supplies
PCR Mix Selection Guide
Protocol
Troubleshooting
Materials
References
Technology Overview: SYBR Green qPCR
With the development of thermal cyclers incorporating fluorescent detection, PCR has a new, innovative application. In routine PCR, the critical result is the final quantity of amplicon generated after the process. Real-time or Quantitative PCR and RT-PCR use the linearity of DNA amplification to determine absolute or relative amounts of a known sequence in a sample. By using a fluorescent reporter in the reaction, it is possible to measure DNA generation.
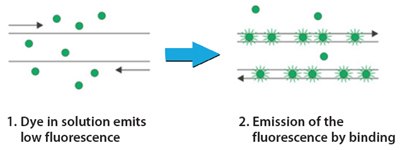
Figure 1.Technology Overview: SYBR Green qPCR
In quantitative PCR, DNA amplification is monitored at each cycle of PCR. When the DNA is in the log linear phase of amplification, the amount of fluorescence increases above the background. The point at which the fluorescence becomes measurable is called the Quantification Cycle (Cq) or crossing point. By using multiple dilutions of a known amount of standard DNA, a standard curve can be generated of log concentration against Cq. The amount of DNA or cDNA in an unknown sample can then be calculated from its Cq value.
A) The different phases of the reaction:
Baseline: The initial concentration of template is low; therefore, the fluorescence intensity is too low to be detected and only the background signal is evident.
Exponential: After the target yield has reached the detection threshold, shown as the red threshold line, the course of the reaction can be followed through the exponential phase.
Linear: As the concentration of template increases, the available DNA polymerase concentration reduces and the reaction rate decreases.
Plateau: There is insufficient free enzyme to continue amplification and so after this point, the reaction is at the maximum yield, or the plateau phase.
B) Individual reactions are characterized by the cycle at which fluorescence first rises above the threshold, which is referred to as the Quantification Cycle (Cq). If the starting material is abundant, amplification is observed in earlier cycles, and the Cq is lower. If the starting material is scarce, amplification is observed in later cycles, and the Cq is higher. This correlation between fluorescence, Cq, and amount of amplified product enables quantification of the template over a wide dynamic range.
Real time PCR also lends itself to relative studies. A reaction may be performed using primers unique to each region to be amplified and tagged with different fluorescent dyes. Several commercially available quantitative thermal cyclers include multiple detection channels. In this multiplex system, the amount of target DNA/cDNA can be compared to the amount of a housekeeping sequence e.g. GAPDH or β-actin.
NR= not recommended
X= recommended
XX= preferred method
Assay Considerations
DNA Preparation
The single most important step in assuring success with PCR is high-quality DNA preparation. Integrity and purity of DNA template are essential. Quantitative PCR involves multiple rounds of enzymatic reactions and is, therefore, more sensitive to impurities such as proteins, phenol/chloroform, salts, EDTA, and other chemical solvents. Contaminants can also interfere with fluorescence detection. The ratio of absorbance values at 260 nM and 280 nM gives an estimate of DNA purity. Pure DNA has an A260/A280 ratio of 1.8-2.0. Lower ratios indicate the presence of contaminants such as proteins.
Template
Very few copies of target nucleic acid (equivalent to about 100 pg of gDNA or cDNA) are needed to initiate qPCR. To minimize contamination with reaction inhibitors, the starting template amount should be kept to the minimum required to achieve accurate quantification. When the starting material is RNA, primer design and DNase I treatment will reduce signals that may be generated from gDNA contamination.
Primer Design
Whether using a dsDNA-binding dye or a probe-based detection chemistry, designing high-quality primers is one of the most crucial pre-experimental steps in qPCR. Specific primers for PCR should be designed with the aid of primer design software to eliminate the complications introduced with primer-dimers and secondary structures. Lower primer concentrations decrease the accumulation of primer-dimer formation and nonspecific product formation, which is critical in using SYBR Green I dye in quantitative PCR.
dNTPs
Standard PCR/qPCR mastermixes contain dATP, dCTP, dGTP, and dTTP. However, some mixes are available that replace dTTP with dUTP. Products from previous reactions run with dUTP will contain uracil instead of thymine. These are then susceptible to cleavage by Uracil-DNA-Glycosylase (UNG). Therefore, prior incubation of subsequent reactions with UNG prevents carry-over contamination between reactions. To be effective, all reactions in the laboratory must use dUTP.
Magnesium Concentration
Magnesium chloride (MgCl2) is necessary for reverse transcriptase, Taq DNA polymerase, and Taq DNA 5’ to 3’ exonuclease activity. Optimum Mg2+ concentrations for reactions containing DLP are usually between 3 – 6 mM. Lower magnesium chloride concentrations usually result in the formation of fewer nonspecific products. Some ReadyMix solutions are provided at a 2X concentration of 7 mM magnesium chloride (final concentration 3.5 mM). In some cases, a vial of a 25 mM magnesium chloride solution is provided for further optimization of the final magnesium chloride concentration if necessary. A reaction mix that does not contain MgCl2 may sometimes be required so that a low concentration can be used, e.g. when using Scorpion Probe detection.
Reverse Transcriptase
A reverse transcriptase enzyme that provides high yields of cDNA, while retaining activity at high temperature, is critical to the success of RT-qPCR. Performance at high temperatures helps to ensure that regions of RNA with significant secondary structure are destabilized and accessible for hybridization and subsequent amplification. When performing one-step RT-qPCR, high-temperature performance allows the use of gene-specific primers with high melting temperatures (Tm), which increases reaction specificity. When performing two step protocols, it is important to ensure that the enzyme results in a linear and proportional yield of cDNA from RNA. Minimizing pipetting can decrease variability. Some ReadyMixes contain primers and other reagents needed to perform RT, for example, ReadyScript® cDNA Synthesis Mix (RDRT).
Taq DNA Polymerase
As with selecting the most appropriate reverse transcriptase for the RT, selection of the appropriate enzyme is vital. A fundamental problem with natural Taq DNA polymerase is that the enzyme has residual activity at low temperature. Non-specific primer binding leads to non-specific product formation as a result of this residual polymerase activity. Antibody-blocked or chemically-blocked Taq DNA polymerases (‘hot-start’) help to rectify this situation by preventing enzyme activity until the high-temperature, denaturation step begins. Refer to the PCR Mix Selection Guide to define the best hot-start polymerase for your application.
Internal Reference Dye by Instrument Type
Some real-time PCR thermal cyclers require a loading dye such as ROX to control for variability in the optical system and to normalize differences in signal intensity. Likewise, some thermal cyclers require fluorescein to create a virtual background when working with SYBR Green I dye assays (which have very low background). These may be supplied in the ReadyMix or as separate components so the appropriate concentration can be used. In some cases, a vial of internal reference dye is included for reaction normalization. Maximum excitation of this dye is 586 nM and maximum emission is 605 nM. Standard instrument settings for ROX reference dye are satisfactory for the measurement of the internal reference dye. This internal reference dye is necessary for ABI Sequence Detection Systems.
Instruments
Reagents compatible with instruments will need to be selected. Platforms use different normalization dyes, so reagents with compatible normalization dyes will need to be selected (refer to Appendix 1).
Many qPCR instruments have been designed to support a specific range of applications, e.g. contrast the ABI 7900 high throughput capability using automatic loading of 384-well plates with the Illumina Eco instrument that supports a single 48-well plate. The most suitable instrument meets the needs of the research. It is desirable to select an instrument with user friendly software that performs the most desirable functions and has flexibility in terms of data output so that it can easily be manipulated in downstream statistical analysis software packages. This reduces the time required to train personnel and therefore to begin generating results. Additional features that are required include a PCR block that is absolutely uniform (an absolute maximum deviation of 1Cq = 2 fold across 96 wells of replication) and an optical system that excites and detects emission as sensitively and as evenly as possible across a wide range of wavelengths. This allows for a wide choice of fluorophores and enables multiplexing. Other features to consider are the operating costs associated with specific consumables, e.g. if a standard microtiter plate is not used for reactions and also the convenience of loading plates/tubes that are non-standard format.
Controls
A positive control is always helpful to make sure all of the kit components are working properly. A no template/negative control is necessary to determine if contamination is present. A signal in the no template control demonstrates the presence of DNA contamination or primer dimer formation.
Buffer
Buffers or reaction mastermixes typically contain dNTPs, a Taq DNA polymerase, MgCl2, and stabilizers. SYBR Green I, ROX™, fluorescein, and inert loading dyes may also be included, depending on the detection chemistry, instrument, and reaction requirements. The PCR buffer components and stabilizers are typically proprietary to the manufacturer. If purchased separately, maximum flexibility is possible, since each ingredient can be individually optimized in the reaction. However, in contrast, while purchasing the ingredients together as a mastermix reduces flexibility, it increases batch consistency and convenience while reducing the number of pipetting steps, and hence, the chances of error and contamination.
Data Analysis
Follow the recommendations of the real time instrument used to perform quantitative SYBR Green PCR. The following may help new instrument users. Generally the number of cycles is plotted against the fluorescence. Threshold cycles (CTs) or crossing points are used to determine the template amount in each sample. Threshold cycle or crossing point is the first cycle that shows a detectable increase in fluorescence due to the formation of PCR products. The cycles before the crossing point are the baseline cycles. The baseline cycles show no detectable increase in fluorescence due to PCR products. The threshold used to determine when the first detectable increase in fluorescence occurs may also be adjusted manually. The threshold should always be done on a logarithmic amplification plot. In a logarithmic amplification plot the threshold should be set in the log-linear range and not the plateau phase.
Melting Curves
Performing a melting curve analysis at the end of the run will help to analyze only the PCR product of interest. Follow the real time instrument manufacturer’s instructions for melting curve analysis. Successive runs with the same primers can be modified to remove the contribution of primer dimer formation to product signal by collecting data in an additional cycling step, the temperature of which must lie between the already determined dimer and product melting temperatures (TMs).
Methods of Quantification
Standard Curves
Standard curves are necessary for both absolute and relative quantification. When generating standard curves, different concentrations of DNA (typically five) should be used to generate a standard curve that will bracket the concentration of the unknown. Each concentration should be run in duplicate.
Absolute and Relative Quantification
This SYBR Green PCR kit may be used to quantify target DNA using either absolute or relative quantification. Absolute quantification techniques are used to determine the amount of target DNA in the initial sample, while relative quantification determines the ratio between the amount of target DNA and a reference amplicon. The ideal reference amplicon would have invariant, constitutive expression. In practice, a housekeeping gene is chosen for this function, but there are other reference choices which better adhere to the above requirements.1
Absolute quantification uses external standards to determine the absolute amount of target nucleic acid of interest. To remove the differences in quantification due to annealing, the primer binding sites of the external standards must be the same as those in the target sequence. The ideal external standard contains sequences that are the same as the target sequence or which vary only slightly from the target sequence. Equivalent amplification efficiencies between the target and external standard are necessary for absolute quantification. Once a suitable construct or amplicon is identified, a standard curve of external standard dilutions is generated and used to determine the concentrations of unknown target samples.
Relative quantification allows calculation of the ratio between the amount of target template and a reference template in a sample. Since this method measures the amount of target relative to a presumably invariant control, relative qPCR is most often used to measure genetic polymorphism differences, for instance, between tissues or between healthy and diseased samples. The advantage of this technique is that using an internal standard can minimize the variations in sample preparation and handling. When using SYBR systems, the target and internal reference quantification must be run in separate reactions.
The accuracy of relative quantification depends on the appropriate choice of a reference template for standards. Variability of the standard will influence the results and so it is most important that standards be appropriate.1 Some researchers choose not to run a standard curve and report target quantities as a fraction of the reference, a technique termed comparative quantitation. Alternatively, one may assume that the amplification efficiencies of target and reference are negligible and quantify target based solely on the standard curve determined for the reference sequence. Finally, in the most accurate of the relative quantification techniques, the amplification efficiencies of both the reference and target are measured, and a correction factor is determined. This process, termed normalization,1 requires a sample containing known concentrations of both target and reference and the generation of two standard curves.
Determination of PCR Reaction Efficiencies
The PCR efficiency between a reference sample and a target sample is determined by preparing a dilution series for each target. The CT values of the reference are subtracted from the target and this difference in CT values is plotted against the logarithm of the template amount. If the resulting slope of the straight line is less than ± 0.1 the amplification efficiencies are judged to be similar.
Equipment
- Quantitative PCR instrument
- Microcentrifuge
- Laminar flow hood for PCR set up (optional)
Supplies
- qPCR SYBR Green Mix – Refer to qPCR Selection Guides (Part 1 and Part 2)
- DNA/cDNA template— cDNA reaction diluted 1:10 to detect a medium to highly expressed targets or 1:2 to 1:5 for rare transcripts or 10 ng to 100 ng gDNA
- Forward and reverse primers diluted to working concentration (10µM working stocks are sufficient for most assays)
- Predesigned gene expression primers are also available for most model organisms (KiCqStart® SYBR® Green Primers, KSPQ12012)
- Sterile filter pipette tips
- Sterile 1.5 mL screw-top microcentrifuge tubes (such as CLS430909)
- PCR tubes, select tubes to match desired format:
- PCR grade water (W1754)
Protocol
Preparation
- Place all reaction components on ice.
- Mix and then briefly centrifuge to collect contents at the bottom of the tube.
Standard SYBR Green I Dye Reaction
Note: We have observed that assays run in KiCqStart ReadyMix are optimal when using a higher primer concentration than in conventional PCR. In the protocols below, we use 450 nM final concentration which we have observed to be the optimal concentration for several independent assays.
1. Prepare enough master mix to run all samples in duplicate.
a. Be sure to include duplicate No template Negative Controls (NTC).
b. Select appropriate table below based upon qPCR reagent selected.
c. Calculate amount of reagents to mix. Add 10% volume to allow for pipetting error
d. Mix well, avoiding bubbles.
Master Mix for KiCqStart Reagents:
Master Mix for Other Complete qPCR ReadyMixes:
Master Mix for qPCR Reagents with Separate Components:
1Optimal MgCl2 concentration can range from 1mM to 6mM.
2Refer to Appendix 1 to determine optimal reference dye concentration for your instrument.
2. Setup reactions:
a. For NTC reactions, add 4 μL of water to the reaction tube.
b. For experimental reactions, add 4 μL of cDNA solution to the reaction tube.
c. Centrifuge all tubes briefly. Visually confirm that all tubes or wells contain sample at the bottom at the correct volume.
d. Carefully aliquot 16 μL of template master mix into each qPCR tube or plate well.
e. Mix reactions well and spin if needed.
f. Cap tubes or seal the PCR plate and label (according to instrument requirements). (Make sure the labelling does not obscure instrument excitation/detection light path.)
3. Run samples as per instrument manufacturer recommendations. Examples of standard and fast cycling have been included below.
Standard Cycling Parameters:
Fast Cycling Parameters:
Troubleshooting Guide
Materials
To continue reading please sign in or create an account.
Don't Have An Account?